 Репликация ДНК: двойная спираль распаковывается и разматывается, затем каждая отделенная нить (бирюза) действует как шаблон для репликации новой партнерской нити (зеленая). Нуклеотиды (основания) подбираются для синтеза новых партнерских цепей в две новые двойные спирали.
Репликация ДНК: двойная спираль распаковывается и разматывается, затем каждая отделенная нить (бирюза) действует как шаблон для репликации новой партнерской нити (зеленая). Нуклеотиды (основания) подбираются для синтеза новых партнерских цепей в две новые двойные спирали. В молекулярной биологии, репликация ДНК - это биологический процесс получение двух идентичных копий ДНК из одной исходной молекулы ДНК. Репликация ДНК происходит во всех живых организмах, выступая в качестве выступая в качестве части части. Клетка отличительным своим делением, что делает репликацию необходимой ДНК.
ДНК состоит из двойной спирали двух дополнительных цепей. Во время репликации эти нити разделяются. Каждая нить исходной молекулы ДНК служит шаблоном для производства своего аналога, процесс, называемый полуконсервативной репликацией. В результате полуконсервативной репликации новая спираль будет состоять из исходной цепи ДНК, а также из вновь синтезированной цепи. Клеточные механизмы проверки и проверки обеспечивают почти идеальную точность репликации ДНК.
В клетке репликация ДНК начинается в определенных местах, или источники репликации в геноме. Раскручивание ДНК в ориджине и синтез новых цепей, аккомодированных ферментом, известным как геликаза, приводит к репликационным вилкам, растущим в двух направлениях от начала. Ряд белков связаны с репликационной вилкой, чтобы помочь в инициации и продолжении синтеза ДНК. Наиболее заметно, что ДНК-полимераза синтезирует новые цепи путем добавления нуклеотидов, которые дополняют каждую (матричную) цепь. Репликация ДНК происходит во время S-стадии интерфазы.
. Репликация ДНК (амплификация ДНК) также может быть выполнена in vitro (искусственно, вне клетки). ДНК-полимеразы, выделенные из клеток, и искусственные праймеры ДНК могут быть использованы для запуска ДНК-ДНК последовательностей в матричной молекуле ДНК. Полимеразная цепная реакция (ПЦР), лигазная цепная реакция (LCR) и транскрипционная амплификация (TMA) являются примерами.
ДНК существует в виде двухцепочечной структуры, в которой обе цепи скручены вместе с образованием характерной двойной спирали. Каждая отдельная цепь ДНК представляет собой цепь из четырех типов нуклеотидов. Нуклеотиды в ДНК содержат дезоксирибозу, сахар, фосфат и азотистое основание. Четыре типа нуклеотида соответствуют четырем нуклеотидным основаниям аденин, цитозин, гуанин и тимин, обычно обозначаемые как A, C, G и T. Аденин и гуанин являются пуриновыми основаниями, а цитозин и тимин - пиримидинами. Эти нуклеотиды образуют фосфодиэфирные связи, создавая фосфат-дезоксирибозный остов двойной спирали ДНК с азимутальными основаниями, направленными внутрь (то есть в сторону противоположной цепи). Нуклеооснования сопоставляются между цепями посредством водородных связей образование пар оснований. Аденин соединяется с тимином (водородные связи), а пары гуанина с цитозином (три водородные связи ).
Цепи ДНК имеют направленность, и разные концы одной цепи называются «3 '(трехпозиционным) концом» и «5' (пятиконечным) концом». По соглашению, если дана последовательность оснований одной цепи ДНК, левый конец придерживается 5'-концом, а правый конец придерживается - 3'-концом. Нити двойная спирали антипараллельны, одна от 5 'до 3', а противоположная нить от 3 'до 5'. Эти термины термины к атому углерода в дезирибозе, к которому присоединяется следующий фосфат в цепи. Направленность имеет последствия для синтеза ДНК, потому что ДНК-полимераза может синтезировать ДНК только в одном направлении, добавляя нуклеотиды к 3'-концу цепи ДНК.
Спаривание комплементарных оснований в ДНК (посредством водородных связей ) означает, что информация, содержащаяся в каждой цепи, является избыточной. Фосфодиэфирные (внутрицепочечные) связи прочнее, чем водородные (межцепочечные). Это позволяет отделить пряди друг от друга. Следовательно, нуклеотиды на одной цепи можно использовать для реконструкции нуклеотидов на вновь синтезированной цепи-партнере.
 ДНК-полимераза нуклеотидов к 3'-концу цепи ДНК. Если несоответствие случайно включено, дальнейшее удлинение полимеразы ингибируется. Проверка удаляет несовпадающий нуклеотид, и удлинение продолжается.
ДНК-полимераза нуклеотидов к 3'-концу цепи ДНК. Если несоответствие случайно включено, дальнейшее удлинение полимеразы ингибируется. Проверка удаляет несовпадающий нуклеотид, и удлинение продолжается. ДНК-полимеразы обеспечивающие собой семейство ферментов, осуществляющих все формы репликации ДНК. ДНК-полимеразы в целом не может инициировать синтез новых цепей. Чтобы начать синтез, необходимо создать короткий фрагмент РНК, называемый праймером, и спарить его с цепью ДНК-матрицы.
ДНК-полимераза новой цепи ДНК, удлиняя 3'-конец существующей нуклеотидной цепи, добавляя новые нуклеотиды, совпадающие с цепочкой-матрицей, по одному за один раз путем создания фосфодифирные связи. Энергия для этого процесса полимеризации ДНК поступает от гидролиза высокоэнергетических фосфатных (фосфоангидридных) связей между тремя фосфатами, присоединенными к каждому невключенному основанию. Свободные основания с присоединенными к ним фосфатными группами называются нуклеотидами ; в частности, основания с тремя присоединенными фосфатными группами называются нуклеозидтрифосфатами. Когда нуклеотид добавляется к растущей цепи ДНК, образование фосфодиэфирной связи между проксимальным фосфатом нуклеотида и растущей цепью сопровождается гидролизом высокоэнергетической фосфатной связи с высвобождением двух дистальных фосфатов в виде пирофосфат. Ферментативный гидролиз полученного пирофосфата в неорганический фосфат потребляет вторую высокоэнергетическую фосфатную связь и делает реакцию необратимой.
В целом ДНК-полимеразы очень точны, с точностью до одной ошибки на каждые 10 добавленных нуклеотидов. Кроме того, некоторые ДНК-полимеразы обладают способностью корректировать; они могут удалять нуклеотиды с конца растущей нити, чтобы исправить несовпадающие основания. И, наконец, механизмов несоответствует ДНК на предмет ошибок цепи. Вместе эти шага различения обеспечивают точность репликации одной ошибки на каждые 10 добавленных нуклеотидов.
Скорость репликации ДНК в живой клетке сначала измерялась как скорость удлинения ДНК фага Т4 в инфицированных фагом E. coli. В период экспоненциального увеличения ДНК при 37 ° C скорость составила 749 нуклеотидов в секунду. Частота мутаций на пару оснований на репликацию во время синтеза ДНК. Фага Т4 составляет 1,7 на 10.
 Обзор этапов репликации ДНК
Обзор этапов репликации ДНК  Этапы ДНК
Этапы ДНК Репликация ДНК, например Все процессы биологической полимеризации протекают в три скоординированных и катализируемых ферментами стадии: инициация, удлинение и прекращение.
 Роль инициаторов в инициации репликации ДНК.
Роль инициаторов в инициации репликации ДНК. 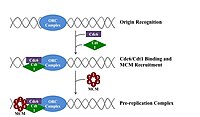 Формирование пререпликационного комплекса.
Формирование пререпликационного комплекса. клетка делилась, она должна сначала реплицировать свою ДНК. Репликация ДНК - это процесс «все или ничего»; как только репликация начинается, она продолжается до завершения. После завершения репликации она больше не повторяется в том же клеточном цикле. Это стало возможным благодаря разделению инициации пререпликационного комплекса.
Пререпликационного комплекса
В позднем митозе и ранней фазе G1 большой Комплекс белков-инициаторов собирается в пререпликационный комплекс в точках ДНК, как известные ориджины ». В Э. coli, первичным белком-инициатором является DnaA ; в дрожжах это комплекс распознавания происхождения . Последовательности, используемые белками-инициаторами, имеют тенденцию быть «AT-богатыми» (богатыми основаниями аденина и тимина), потому что пары оснований AT имеют две водородные связи (а не три, образованные в паре CG), и, следовательно, их легче разделить на цепи. У эукариот комплекс распознавания ориджина катализирует сборку белков-инициаторов в пререпликационный комплекс. Cdc6 и Cdt1 затем связываются со комплексом распознавания в источнике, чтобы сформировать более крупный комплекс, необходимый для загрузки комплекса Mcm на ДНК. Комплекс Mcm - это геликаза, которая распутывает спираль ДНК в точках начала репликации и вилках репликации у эукариот. Комплекс Mcm рекрутируется на поздней фазе G1 и загружается комплексом ORC-Cdc6-Cdt1 на ДНК посредством АТФ-зависимого ремоделирования белка. Нагрузка комплекса Mcm на исходную ДНК знаменует завершение образования пререпликационного комплекса.
Если условия окружающей среды подходят к поздней фазе G1, G1 и G1 / S циклин - Комплексы Cdk активируют, что стимулирует экспрессию генов, кодирующие компоненты аппарата ДНК. Активация G1 / S-Cdk также экспрессии активации комплексов S-Cdk, которые могут играть роль в активации систем репликации в зависимости от вида и типа клеток. Контроль этих CDK зависит от типа клетки и стадии развития. Эта регуляция лучше всего понятна у почкующихся дрожжей, где S-циклины Clb5 и Clb6 в первую очередь соответствуют за репликацию ДНК. Комплексы Clb5,6-Cdk1 непосредственно запускают активацию ориджинов репликации и, следовательно, необходимы на протяжении S-фазы для прямого запуска каждого ориджина.
Аналогичным образом Cdc7 также требуется через S-фаза для активации источников репликации. Cdc7 не активен на протяжении клеточного цикла, и его активация строго рассчитана по времени, чтобы избежать преждевременного запуска репликации ДНК. В конце G1 активность Cdc7 резко возрастает в результате ассоциации с регуляторной субъединицей Dbf4, которая напрямую связывает Cdc7 и способствует его активности протеинкиназы. Было обнаружено, что Cdc7 является ограничивающим регулятором происхождения скорости. Вместе с G1 / S-Cdks и / или S-Cdks и Cdc взаимодействуют, чтобы напрямую активировать источники репликации, что приводит к инициации синтез ДНК.
Комплекс преинициации
В ранней активации S-фазы S-Cdk и Cdc7 вызывают сборку преинициативного комплекса, массивного белкового комплекса, образованного в начале. Формирование преинициативного комплекса вытесняет Cdc6 и Cdt1 из исходного репликационного комплекса, инактивируя и разобирая пре-репликационный комплекс. Загрузка преинициативного комплекса в ориджин активирует геликазу Mcm, вызывая раскручивание спирали ДНК. Комплекс преинициирования также загружает α-примазу и другие ДНК-полимеразы в ДНК.
После того, как α-примаза синтезирует первые праймеры, соединения праймер-матрица взаимодействуют с загрузчиком зажимов, который загружает скользящий зажим на ДНК, чтобы начать синтез ДНК. Компоненты преинициативного комплекса остаются связанными с репликационными вилками по мере их удаления от источника.
ДНК-полимераза обладает 5'– 3 'активностью. Все известные системы ДНК требуют наличия 3 'гидроксильной группы, прежде чем можно будет инициировать синтез репликации: матрица ДНК считается в направлении от 3' до 5 ', как новая цепь в направлении 5' до 3 '. 'Направление - это часто путают). Известны четыре различных механизма ДНК:
Первый - это наиболее известный из этих механизмов и используемых мобильных устройств. В этом механизме после разделения двух цепей примаза перемеры РНК к цепям матрицы. Ведущая цепь получает один праймер РНК, а отстающая цепь - несколько. Ведущая цепь непрерывно удлиняется от праймера с помощью ДНК-полимеразы с высокой процессивностью, в то время как отстающая цепь продолжается прерывисто от каждого праймера, образуя фрагменты Окадзаки. РНКаза удаляет фрагменты праймерной РНК, входит ДНК-полимераза с низкой процессивностью, отличная от репликативной полимеразы, чтобы заполнить пробелы. Когда это будет завершено, можно будет найти одну щель на ведущей нити и несколько неровностей на отстающую нити. Лигаза заполняет эти разрывы, завершая реплицируемую молекулу ДНК.
Примаза, используемая в этом процессе, особенно различается между бактериями и архей / эукариотами. Бактерии используют примазу, принадлежащую к суперсемейству белков DnaG, которая содержит каталитический домен складчатого типа TOPRIM. Сгиб TOPRIM содержит ядро α / β с четырьмя консервативными цепями в топологии Россманна. Эта структура также обнаруживается в каталитических доменах топоизомеразы Ia, топоизомеразы II, нуклеаз семейства OLD и белков репарации ДНК, связанных с белком RecR.
Примаза, используемая археями и эукариотами, напротив, содержит высокопроизводительную версию мотива распознавания РНК (RRM). Эта праймаза структурно сходна со многими вирусными РНК-зависимыми РНК-полимеразами, обратными транскриптазами, циклами, генерирующими циклические нуклеотиды, ДНК-полимеразами семейств A / B / Y, которые участвуют в репликации и репарации ДНК. При репликации эукариот праймаза образует комплекс с Pol α.
Множественные ДНК-полимеразы играют разные роли в процессе репликации ДНК. В Э. coli, ДНК Pol III представляет собой фермент-полимеразу, который в первую очередь отвечает за репликацию ДНК. Он собирается в течение всего цикла репликации, оставаясь нетронутым в течение всего цикла репликации. Напротив, ДНК Pol I представляет собой фермент, отвечающий за замену праймеров РНК на ДНК. ДНК Pol I обладает 5'-3'-экзонуклеазной активностью в дополнение к своей полимеразной активности и использует свою экзонуклеазную активность для разрушения праймеров РНК впереди себя, поскольку он удлиняет цепь ДНК позади себя, в процессе называется ник перевод. Pol I намного менее процессивен, чем Pol III, потому что его основная функция в множестве коротких участков ДНК, а несколько не очень длинных участков.
У эукариот фермент с низкой процессивностью, Pol α инициирует репликацию, поскольку он образует комплекс с примазой. У эукариот синтез ведущей цепи, как полагают, осуществляется Pol ε; однако это мнение недавно было оспорено, предполагаемая роль Пол δ. Удаление праймера завершается Pol δ, а восстановление ДНК во время репликации завершается Pol ε.
По мере продолжения синтеза ДНК исходные цепи ДНК продолжают раскручиваться с каждой стороны пузырька, образуя репликационную вилку с двумя зубцами. У бактерий, которые имеют единственную точку начала репликации на своей кольцевой хромосоме, этот процесс создает «тета-адаптированная » (напоминающая греческую букву тета: θ). Напротив, эукариоты имеют более длинные линейные хромосомы и инициируют репликацию в нескольких источниках внутри них.
 Схема репликационной вилки.. a :, b: ведущая цепь, c: отстающая цепь, d: репликационная вилка, e: праймер, f: фрагменты Окадзаки шаблон
Схема репликационной вилки.. a :, b: ведущая цепь, c: отстающая цепь, d: репликационная вилка, e: праймер, f: фрагменты Окадзаки шаблон  Многие ферменты участвуют в репликационной вилке ДНК.
Многие ферменты участвуют в репликационной вилке ДНК. Репликационная вилка - это структура, которая формируется внутри длинной спиральной ДНК во время репликации ДНК. Он создает геликазами, которые разрывают водородные связи, удерживают две нити ДНК вместе в спирали. Полученная структура имеет два ветвящихся «зубца», каждый из которых состоит из одной нити ДНК. Эти две цепи в качестве матрицы для ведущей цепи, которая создается, когда ДНК-полимераза сопоставит комплементарные нуклеотиды шаблонам; шаблоны могут правильно называться шаблоном ведущей нити и шаблоном отстающей нити.
ДНК-ДНК-полимеразой в направлении от 3 'до 5', что означает, что считающая цепь синтезируется в направлении от 5 'до 3'. матрица ведущая и отстающая вилка цепи ориентирована в противоположных направлениях в направлении Репликационная вилка, основная проблема заключается в том, как возникает зарождающаяся (новая) отстающей цепи цепи, направление противоположно противоположной растущей репликационной вилки.
Ведущая цепь - это цепь зарождающейся ДНК, которая синтезируется в том же направлении, что и растущая репликационная вилка. Этот вид репликации ДНК непрерывен.
Отстающая нить - это нить растущей ДНК, направление противоположно противоположно растущей репликационной вилки. Из-за своей ориентации репликация отстающей цепи более сложная по сравнению с репликацией ведущей цепи. Как следствие, ДНК-полимераза на этой цепи, как видно, «отстает» от другой цепи.
Отстающая нить синтез в виде коротких отдельных сегментов. Направление отстающей цепи примаза «считывает» матричную ДНК и синтезирует синтез короткого комплементарного праймера РНК. ДНК-полимераза удлиняет примированные сегменты, образуя фрагменты Окадзаки. Затем праймеры РНК удаляются и заменяются ДНК, фрагменты ДНК объединяются с помощью ДНК-лигазы.
 Собранный зажим ДНК человека, тример белка PCNA.
Собранный зажим ДНК человека, тример белка PCNA.Во всех случаев геликаза состоит из шести полипептидов, которые охватывают только одну цепь реплицируемой ДНК. Две полимеразы связаны с гексимером геликазы. У эукариот геликаза оборачивается вокруг ведущей цепи, а у прокариот - вокруг отстающей цепи.
Когда геликаза раскручивает ДНК на репликационной вилке, ДНК впереди заставляет вращаться. Этот процесс приводит к накоплению изгибов в ДНК впереди. Это нарастание образует сопротивление скручиванию, которое в конечном итоге остановит продвижение репликационной вилки. Топоизомеразы - это ферменты, которые временно разрывают цепи ДНК, снимаемое напряжение, вызванное раскручиванием двух цепей спирали ДНК; топоизомеразы (включая ДНК-гиразу ) достигают этого путем добавления отрицательных суперспиралей к спирали ДНК.
Открытая одноцепочечная тенденция складываться обратно, образуя второстепенные конструкции ; эти структуры мешают движению ДНК-полимеразы. Чтобы предотвратить это, одноцепочечные связывающие белки связываются с ДНК до тех пор, пока не будет синтезирована вторая цепь, предотвращающая образование вторичной структуры.
Двухцепочечная ДНК наматывается вокруг гистонов, которые играют роль в регуляции экспрессии генов, поэтому реплицированная ДНК должна быть намотана вокруг гистонов в тех же местах, что и исходная ДНК. Чтобы это гистоновые шапероны разбирают хроматин перед репликацией и заменяют гистоны в нужном месте. Некоторые шаги в этой сборке несколько спекулятивны.
Зажимные белки через скользящий зажим вокруг ДНК, помогая ДНК-полимеразе поддерживать контакт со матрицей, самым своим способом процессивности. Внутренняя поверхность поверхности позволяет пропустить через него ДНК. Как только полимераза достигает конца матрицы или обнаруживаетцепочечную ДНК, скользящий зажим претерпевает конформационное изменение, высвобождает ДНК-полимеразу. Белки, нагружающие зажимы, используются для предварительной нагрузки зажима, распознавая соединение между матрицей и праймерами РНК.
На вилке репликации многие репликации ферментации собираются ДНК в сложной молекулярной машине, названная реплисомой. Ниже представлен список основных ферментов репликации ДНК, которые участвуют в реплисоме:
| Фермент | Функция репликации ДНК |
|---|---|
| ДНК-геликаза | Также известен как фермент, дестабилизирующий спираль. Геликаза разделяет две нити ДНК на вилке репликации за топоизомеразой. |
| ДНК-полимераза | Фермент, ответственный за катализирование добавления времени нуклеотидных субстратов к ДНК в направлении от 5 'к 3' во репликации ДНК. Также корректуру и исправление ошибок. Существует много разных типов ДНК-полимеразы, каждая из которых выполняет разные функции в разных типах клеток. |
| Зажим ДНК | Белок, который предотвращает диссоциацию удлиняющих ДНК-полимераз от родительской цепи ДНК. |
| Одноцепочечный ДНК-связывающий белок | Связывается с оцДНК и предотвращает повторный отжиг двойной спирали ДНК после того, как ДНК-геликаза раскручивает ее, таким образом поддерживая разделение цепей и облегчая синтез формирующейся цепи. |
| Топоизомераза | Освобождает ДНК от ее суперспиральной природы. |
| ДНК-гираза | Снимает напряжение раскручивания ДНК-геликазой; это особый тип топоизомеразы |
| ДНК-лигаза | Повторно отжигает полуконсервативные цепи и соединяет фрагменты Окадзаки отстающей цепи. |
| Примаза | Обеспечивает точку РНК (или ДНК) для ДНК-полимеразы, чтобы начать синтез новой цепи ДНК. |
| Теломераза | Удлиняет теломерную ДНК за счет добавления повторяющихся нуклеотидных последовательностей к концам эукариотических хромосом. Это позволяет зародышевым клеткам и стволовымкам избежать ограничения Хейфлика на деление клеток. |
 E. coli Replisome. Примечательно, что ДНК на отстающую цепи образует петлю. Точная структура реплисомы не совсем понятна.
E. coli Replisome. Примечательно, что ДНК на отстающую цепи образует петлю. Точная структура реплисомы не совсем понятна. Механизмы репликации состоят из факторов, участвующих в репликации ДНК и появляющихся матричных оцДНК. Механизмы репликации включают примосоторы - ферменты репликации; ДНК-полимераза, ДНК-геликазы, ДНК-зажимы и ДНК-топоизомеразы, а также белки репликации; например одноцепочечные ДНК-связывающие белки (SSB). В механизмх репликации эти компоненты координируются. У некоторых бактерий все факторы, участвующие в репликации ДНК, установлены на вилках репликации, а комплексы остаются на вилках во время репликации ДНК. Эти репликационные устройства называются реплисомами или системами ДНК-репликазы . Эти термины являются общими для белков, используемых на вилках репликации. В эукариотических и некоторых бактериальных клетках реплисомы не образуются.
Механизмы репликации не перемещаются относительно шаблонных ДНК, таких как фабрики, они называются фабрикой репликации . Альтернативной фигурерики ДНК подобны проекторам, ДНК подобны кинематографическим фильмам, которые постоянно проходят в проекторы. В модели фабрики репликации, после того, как обе ДНК-геликазы для ведущих цепей и отстестящих цепей загружены в матрицу ДНК, геликазы перемещаются вдоль ДНК-геликазы друг друга. Хеликазы остаются связанными до конца процесса репликации. Питер Мейстер и др. непосредственно наблюдали сайты репликации в почкующихся дрожжах путем мониторинга ДНК-полимеразы , меченной зеленым флуоресцентным белком (GFP) α. Они получили репликацию ДНК пар помеченных локусов, симметрично разнесенных от точки начала репликации, и появилось расстояние между парами значительно уменьшилось со временем. Это открытие предполагает, что механизм репликации ДНК связан с фабриками ДНК. То есть пары фабрик репликации загружаются в источники репликации и фабрики, связанные друг с другом. Кроме того, матричные ДНК перемещаются на фабрики, что приводит к экструзии матричных оцДНК и зарождающихся ДНК. Находка Мейстера - первое прямое свидетельство модели фабрики репликации. Последующие исследования показали, что образ ДНК-геликазы используют димеры во многих эукариотических клетках, а механизмы репликации бактерий существуют в одномядерном месте во время синтеза ДНК.
Фабрики репликации выполняют распутывание сестринских хроматид. Распутывание для распределения хроматид по дочерним клеткам после репликации ДНК. Системные хроматиды после репликации ДНК удерживают друг друга кольцами Cohesin, существует единственный шанс для распутывания при репликации ДНК. Установка механизмов репликации в фабрик репликации может повысить успешность репликации ДНК. Если вилки репликации свободно перемещаются в хромосомах, сцепление ядер усугубляется и препятствует митотической сегрегации.
Прекращение требует, чтобы продвижение вилки репликации ДНК было остановлено или заблокировано. Терминация в конкретном локусе, когда она происходит, включает взаимодействие между двумя компонентами: (1) последовательность терминации в ДНК и (2) белком, который связывается с этой последовательностью, чтобы физически остановить репликацию ДНК. У различных видов бактерий это называется сайт-связывающим белком конца репликации ДНК или Тер-белком.
. Бактерии используют кольцевые хромосомы, прекращение репликации происходит, когда две репликационные вилки встречаются друг с другом на противоположном конце родительская хромосома. E. coli регулирует этот процесс использования терминирующих последовательностей, которые при связывании с белком Tus обеспечивают прохождение только одного направления репликационной вилки. В результате репликационные вилки вынуждены всегда встречаться в терминальной области хромосомы.
 Клеточный цикл эукариотических клеток.
Клеточный цикл эукариотических клеток. Внутри эукариот, Репликация ДНК контролируется в контексте клеточного цикла. По мере того, как клетка растет и делится, она проходит стадию клеточного цикла; Репликация ДНК происходит во время фазы S (синтез). Прогресс эукариотической клетки по циклу контролируется контрольными точками клеточного цикла. Прохождение через контрольные точки контролируется с помощью различных взаимодействий между различными белками, включая циклины и циклин-зависимые киназы. В отличие от бактерий, эукариотическая ДНК реплицируется в пределах ядра.
Контрольная точка G1 / S (или контрольная точка рестрикции) регулирует, вступают ли эукариотические клетки в процесс репликации ДНК и последующего деления. Клетки, которые не проходят через эту контрольную точку, остаются на стадии G0 и не реплицируют свою ДНК.
После прохождения контрольной точки G1 / S ДНК должна реплицироваться только один раз в каждом клеточном цикле. Когда комплекс Mcm удаляется от источника, пререпликационный комплекс разрушается. Не может быть загружен в новый комплекс Mcm, пока субъединицы предварительной репликации не будут реактивированы, нельзя использовать один источник репликации дважды в одном и том же клеточном цикле.
Активация S-Cdks в ранней S-фазе стимулирования или ингибирования отдельных компонентов пререпликационного комплекса, предотвращает немедленную повторную сборку. S и M-Cdks продолжают блокировать сборку комплекса до репликации даже после завершения S-фазы, гарантирую, что сборка не может произойти снова, пока вся активность Cdk не будет снижена в позднем митозе.
У почкующихся дрожжей ингибирование сборки вызывается Cdk-зависимым фосфорилированием компонентов пререпликационного комплекса. В начале S-фазы фосфорилирование Cdc6 с помощью Cdk1 вызывает связывание Cdc6 с SCF протеинлигазой убиквитина, что вызывает протеолитическое разрушение Cdc6. Cdk-зависимое фосфорилирование белков Mcm способствует их экспорту из ядра вместе с Cdt1 во время фазы S, предотвращает загрузку новых комплексов Mcm в источнике в течение всего клеточного цикла. Cdk фосфорилирование комплекса репликации ориджина также ингибирует сборку комплекса до репликации. Индивидуального присутствия любого из этих трех механизмов достаточно, чтобы ингибировать сборку пререпликационного комплекса. Однако мутации всех трех белков в одной и той же клетке действительно запускают повторную инициацию во многих источниках репликации в пределах одного клеточного цикла.
В клетках животных белок геминин является ключевым ингибитором пре- сборка комплекса репликации. Геминин связывает Cdt1, предотвращая его связывание с исходным комплексом распознавания. В G1 уровни геминина поддерживаются на низком уровне с помощью APC, который убиквитинирует геминин, чтобы нацелить его на деградацию. Когда геминин разрушается, Cdt1 высвобождается, позволяя ему функционировать в предрепликационной сложной сборке. В конце G1 APC инактивируется, позволяя геминину накапливать и связывать Cdt1.
Репликация хлоропластных и митохондриальных геномов происходит независимо от клеточного цикла в процессе репликации D-петли.
В клетках позвоночных сайты репликации концентрируются в положениях, называемых фокусами репликации . Сайты репликации могут быть обнаружены путем иммуноокрашивания дочерних цепей и ферментов репликации и мониторинга факторов репликации, меченных GFP. С помощью этих методов обнаружено, что фокусы репликации различного размера и положения появляются в S-фазе деления клетки, и их количество на ядро намного меньше, чем количество геномных вилок репликации.
П. Heun et al., (2001) отслеживали фокусы репликации, меченные GFP, в почкующихся дрожжевых клетках и обнаружили, что точки начала репликации постоянно перемещаются в фазе G1 и S, а динамика значительно снижается в фазе S. Традиционно сайты репликации фиксировались на пространственной структуре хромосом с помощью ядерного матрикса или ламинов. Результаты Хойна опровергают традиционные представления о том, что у почкующихся дрожжей нет ламинов, и подтверждают, что источники репликации самоорганизуются и образуют центры репликации.
За счет активации источников репликации, контролируемых пространственно и временно, регулируется образование фокусов репликации. D. A. Jackson и др. (1998) обнаружили, что соседние источники одновременно активируются в клетках млекопитающих. Пространственное сопоставление сайтов репликации приводит к кластеризации вилок репликации. Кластеризация спасает застопорившиеся вилки репликации и способствует нормальному продвижению вилок репликации. Прогресс репликационных вилок тормозится многими факторами; столкновение с белками или комплексами, прочно связывающимися с ДНК, дефицит dNTP, разрывы на матричных ДНК и т. д. Если репликационные вилки останавливаются, а оставшиеся последовательности из остановившихся вилок не реплицируются, дочерние цепи имеют нелицензированные нереплицированные сайты. Нереплицированные сайты на одной родительской цепи удерживают вместе другую, но не дочерние цепи. Следовательно, образующиеся сестринские хроматиды не могут отделиться друг от друга и не могут делиться на 2 дочерние клетки. Когда срабатывают соседние источники и разветвление из одного источника останавливается, разветвление из другого источника осуществляет доступ в направлении, противоположном остановившемуся разветвлению, и дублирует нереплицированные сайты. В качестве другого механизма спасения есть применение неактивных ориджинов репликации, которые лишние ориджины не запускаются при нормальной репликации ДНК.
 Dam метилирует аденин сайтов GATC после репликации.
Dam метилирует аденин сайтов GATC после репликации. Большинство бактерий не проходят четко определенный клеточный цикл, а вместо этого постоянно копируют свою ДНК; во время быстрого роста это может привести к одновременному возникновению нескольких циклов репликации. У E. coli, наиболее охарактеризованных бактерий, репликация ДНК регулируется с помощью нескольких механизмов, включая: гемиметилирование и секвестирование исходной последовательности, соотношение аденозинтрифосфата (АТФ) к аденозиндифосфату. (АДФ) и уровни белка DnaA. Все они контролируют связывание белков-инициаторов с исходными последовательностями.
Таймер E. coli метилирует отслеживает ДНК GATC, синтез ДНК приводит к полуметилированным последовательностям. Эта гемиметилированная ДНК распознается белком SeqA, который связывает и изолирует исходную последовательность; кроме того, DnaA (необходимая для инициации репликации) хуже связывается с гемиметилированной ДНК. В результате вновь реплицированные ориджины не могут инициировать следующий раунд репликации ДНК.
АТФ накапливается, когда клетка находится в богатой среде, запуская репликацию ДНК, когда клетка определенного размера. АТФ конкурирует с АДФ за связывание с DnaA, и комплекс DnaA-ATP вызывает инициировать репликацию. Определенное количество белков DnaA также требуется для репликации ДНК - каждый раз, когда копируется источник, количество сайтов связывания для DnaA удваивается, что требует сокращения большего количества DnaA, чтобы обеспечить еще одну инициацию репликации.
У быстрорастущих бактерий, таких как E. coli, репликация хромосом занимает больше времени, чем деление клетки. Бактерии решают эту проблему, инициируя новый раунд репликации до того, как предыдущий был завершен. Новый раунд репликации сформирует хромосому клетки, которая рождается через два поколения после делящейся клетки. Этот механизм перекрывающиеся циклы репликации.
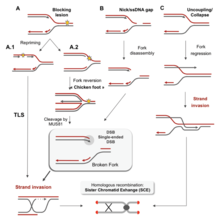 Репликационная вилка перезапускается из-за гомологичной рекомбинации после репликационного стресса
Репликационная вилка перезапускается из-за гомологичной рекомбинации после репликационного стресса 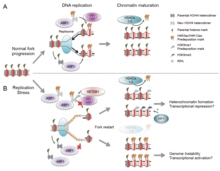 Эпигенетические последствия повторной сборки нуклеосом в остановленных репликационных вилках
Эпигенетические последствия повторной сборки нуклеосом в остановленных репликационных вилках Есть много событий, которые способствуют стрессу репликации, в том числе:
Исследователи обычно реплицируют ДНК in vitro с помощью полимеразной цепной реакции (ПЦР). ПЦР использует пару праймеров для охвата целевой области в матричной ДНК, а затем полимеризует партнерские цепи в каждом направлении от этих праймеров с использованием термостабильной ДНК-полимеразы. Повторение этого процесса через несколько циклов усиливает целевой участок ДНК. В начале цикла смеси матрицы и праймеров нагревают, разделяя вновь синтезированную молекулу и матрицу. Затем, когда смесь остывает, они становятся матрицами для отжига новых праймеров, и полимераза выходит из них. В результате количества копий области удваивается каждый раунд, увеличиваясь экспоненциально.
| Викискладе есть носители, связанные с репликацией ДНК. |
| Викиверситет имеет ресурсы для обучения ДНК # ДНК_Репликация |