 Структура эукариотического гена
Структура эукариотического гена В вычислительной биологии, прогнозирование гена или обнаружение гена относится к процессу идентификации участков геномной ДНК, которые кодируют гены. Это включает в себя кодирующие белки гены, а также гены РНК, но также может включать прогнозирование других функциональных элементов, таких как регуляторные области. Поиск генов - один из первых и наиболее важных шагов в понимании генома вида после того, как он был секвенирован.
На заре своего существования «поиск генов» был основан на кропотливых экспериментах на живых клетках и организмах. Статистический анализ скоростей гомологичной рекомбинации нескольких различных генов может определить их порядок на определенной хромосоме, и информацию из многих таких экспериментов можно объединить для создания генетической карты с указанием примерного расположения известных генов относительно друг друга. Сегодня, когда в распоряжении исследовательского сообщества есть исчерпывающая последовательность генома и мощные вычислительные ресурсы, поиск генов был переосмыслен как в значительной степени вычислительная проблема.
Определение функциональности последовательности следует отличать от определения функции гена или его продукта. Прогнозирование функции гена и подтверждение точности предсказания гена по-прежнему требует экспериментов in vivo с помощью нокаута гена и других анализов, хотя границы биоинформатических исследований остаются что делает все более возможным прогнозирование функции гена только на основе его последовательности.
Прогнозирование гена - один из ключевых шагов в аннотации генома после сборки последовательности, фильтрации некодирующих областей и повторного маскирования.
Прогнозирование генов тесно связано с так называемой «проблемой поиска цели», изучающей, как ДНК-связывающие белки (факторы транскрипции ) находят конкретные сайты связывания в пределах геном. Многие аспекты предсказания структурных генов основаны на текущем понимании основных биохимических процессов в клетке, таких как ген транскрипция, трансляция, белок-белковые взаимодействия и процессы регуляции, которые являются предметом активных исследований в различных областях омики, таких как транскриптомика, протеомика, метаболомика и в более общем плане структурная и функциональная геномика.
В эмпирических (на основе сходства, гомологии или фактических данных) системах поиска генов целевой геном ищется на предмет последовательностей, аналогичных внешним свидетельствам i n форма известных тегов экспрессируемой последовательности, матричной РНК (мРНК), белковых продуктов и гомологичных или ортологичных последовательностей. Учитывая последовательность мРНК, легко получить уникальную последовательность геномной ДНК, из которой она должна была быть транскрибирована. Учитывая последовательность белка, семейство возможных кодирующих последовательностей ДНК может быть получено путем обратной трансляции генетического кода. После определения последовательностей ДНК-кандидатов относительно простой алгоритмической проблемой становится эффективный поиск в целевом геноме совпадений, полных или частичных, точных или неточных. Для данной последовательности алгоритмы локального выравнивания, такие как BLAST, FASTA и Smith-Waterman, ищут области сходства между целевой последовательностью и возможными совпадениями кандидатов. Совпадения могут быть полными или частичными, точными или неточными. Успех этого подхода ограничен содержанием и точностью базы данных последовательностей.
Высокая степень сходства с известной матричной РНК или белковым продуктом является убедительным доказательством того, что область целевого генома является геном, кодирующим белок. Однако для системного применения этого подхода требуется обширное секвенирование мРНК и белковых продуктов. Это не только дорого, но и в сложных организмах в любой момент времени экспрессируется только подмножество всех генов в геноме организма, а это означает, что внешние свидетельства существования многих генов не всегда доступны ни в одной культуре отдельных клеток. Таким образом, для сбора внешних доказательств для большинства или всех генов в сложном организме требуется изучение многих сотен или тысяч типов клеток, что представляет дополнительные трудности. Например, некоторые гены человека могут экспрессироваться только во время развития эмбриона или плода, что может быть трудно изучать по этическим причинам.
Несмотря на эти трудности, были созданы обширные базы данных транскриптов и последовательностей белков для человека, а также других важных модельных организмов в биологии, таких как мыши и дрожжи. Например, база данных RefSeq содержит транскрипт и белковые последовательности из многих различных видов, а система Ensembl всесторонне отображает это свидетельство на человеческий и несколько других геномов. Однако вполне вероятно, что обе эти базы данных неполны и содержат небольшие, но значительные объемы ошибочных данных.
Новые высокопроизводительные технологии секвенирования транскриптома, такие как RNA-Seq и ChIP-секвенирование, открывают возможности для включения дополнительных внешних доказательств в прогнозирование генов и проверка, и позволяют структурно богатую и более точную альтернативу предыдущим методам измерения экспрессии гена, таким как тег экспрессируемой последовательности или ДНК-микрочип.
Основные проблемы, связанные с предсказанием генов связаны с ошибками секвенирования в необработанных данных ДНК, зависимостью от качества сборки последовательности, обработкой коротких чтений, мутациями сдвига рамки считывания, перекрывающимися генами и неполными генами.
У прокариот важно учитывать горизонтальный перенос генов при поиске гомологии последовательностей генов. Дополнительным важным фактором, недостаточно используемым в существующих средствах обнаружения генов, является наличие кластеров генов - оперонов (которые представляют собой функционирующие единицы ДНК, содержащие кластер генов под контролем одного промотора ) как у прокариот, так и у эукариот. Большинство популярных детекторов генов рассматривают каждый ген изолированно, независимо от других, что не является биологически точным.
Прогнозирование гена Ab Initio является внутренним методом, основанным на содержании гена и обнаружении сигнала. Из-за неизбежных затрат и трудностей в получении внешних доказательств для многих генов также необходимо прибегнуть к поиску генов ab initio, в котором геномная последовательность ДНК Одни только систематически ищут определенные контрольные признаки генов, кодирующих белок. Эти признаки можно в широком смысле классифицировать как сигналы, специфические последовательности, указывающие на присутствие гена поблизости, или как содержание, статистические свойства самой последовательности, кодирующей белок. Поиск гена ab initio можно было бы более точно охарактеризовать как прогнозирование гена, поскольку для окончательного установления функциональности предполагаемого гена обычно требуются внешние доказательства.
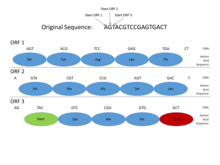 На этом рисунке показано, как открытые рамки чтения (ORF) могут использоваться для прогнозирования генов. Прогнозирование генов - это процесс определения того, где может находиться кодирующий ген в геномной последовательности. Функциональные белки должны начинаться со стартового кодона (где начинается транскрипция ДНК) и заканчиваться стоп-кодоном (где заканчивается транскрипция). Глядя на то, где эти кодоны могут находиться в последовательности ДНК, можно увидеть, где может располагаться функциональный белок. Это важно для прогнозирования генов, потому что может выявить, где находятся кодирующие гены во всей геномной последовательности. В этом примере функциональный белок может быть обнаружен с использованием ORF3, поскольку он начинается со стартового кодона, имеет несколько аминокислот и затем заканчивается стоп-кодоном, все в одной рамке считывания.
На этом рисунке показано, как открытые рамки чтения (ORF) могут использоваться для прогнозирования генов. Прогнозирование генов - это процесс определения того, где может находиться кодирующий ген в геномной последовательности. Функциональные белки должны начинаться со стартового кодона (где начинается транскрипция ДНК) и заканчиваться стоп-кодоном (где заканчивается транскрипция). Глядя на то, где эти кодоны могут находиться в последовательности ДНК, можно увидеть, где может располагаться функциональный белок. Это важно для прогнозирования генов, потому что может выявить, где находятся кодирующие гены во всей геномной последовательности. В этом примере функциональный белок может быть обнаружен с использованием ORF3, поскольку он начинается со стартового кодона, имеет несколько аминокислот и затем заканчивается стоп-кодоном, все в одной рамке считывания. В геномах прокариот, гены имеют специфические и относительно хорошо изученные промоторные последовательности (сигналы), такие как сайты связывания бокса Прибноу и фактора транскрипции , которые легко систематически идентифицировать. Кроме того, последовательность, кодирующая белок, представляет собой одну непрерывную открытую рамку считывания (ORF), которая обычно состоит из многих сотен или тысяч пар оснований в длину. Статистика стоп-кодонов такова, что даже обнаружение открытой рамки считывания такой длины является довольно информативным признаком. (Поскольку 3 из 64 возможных кодонов в генетическом коде являются стоп-кодонами, можно ожидать, что стоп-кодон будет примерно через каждые 20–25 кодонов или 60–75 пар оснований в случайной последовательности.) Кроме того, ДНК, кодирующая белок, имеет определенные периодичности и другие статистические свойства, которые легко обнаружить в последовательности такой длины. Эти характеристики делают поиск прокариотических генов относительно простым, а хорошо спроектированные системы способны обеспечить высокий уровень точности.
Обнаружение гена Ab initio у эукариот, особенно сложных организмов, таких как человек, значительно сложнее по нескольким причинам. Во-первых, промотор и другие регуляторные сигналы в этих геномах более сложны и менее понятны, чем в прокариотах, что затрудняет их надежное распознавание. Два классических примера сигналов, идентифицированных эукариотическими поисковиками генов, - это CpG-островки и сайты связывания для поли (A) хвоста.
Во-вторых, механизмы сплайсинга, используемые эукариотическими клетками, означают что конкретная кодирующая белок последовательность в геноме разделена на несколько частей (экзоны ), разделенных некодирующими последовательностями (интронами ). (Сайты сплайсинга сами по себе являются еще одним сигналом, для идентификации которого часто предназначены эукариотические геноискатели.) Типичный ген, кодирующий белок, у человека можно разделить на дюжину экзонов, каждый из которых имеет длину менее двухсот пар оснований, а некоторые - всего двадцать до тридцати. Следовательно, гораздо труднее обнаружить периодичность и другие известные свойства содержания кодирующей белок ДНК у эукариот.
Продвинутые специалисты по поиску генов как для прокариотических, так и для эукариотических геномов обычно используют сложные вероятностные модели, такие как скрытые марковские модели (HMM), чтобы объединить информацию из множества различных сигналов. и измерения содержания. Система GLIMMER - это широко используемый и высокоточный геноискатель для прокариот. GeneMark - еще один популярный подход. Для сравнения, эукариотические геноискатели ab initio достигли лишь ограниченного успеха; яркими примерами являются GENSCAN и программы. Программа поиска генов SNAP основана на HMM, как и Genscan, и пытается быть более адаптируемой к различным организмам, решая проблемы, связанные с использованием средства поиска генов в последовательности генома, против которой он не был обучен. Некоторые недавние подходы, такие как mSplicer, CONTRAST, или также используют методы машинного обучения, такие как , поддерживают векторные машины для успешного прогнозирования генов. Они создают дискриминантную модель, используя или условные случайные поля, чтобы узнать точную функцию оценки предсказания гена.
Методы Ab Initio были протестированы, при этом чувствительность некоторых приближалась к 100%, однако по мере увеличения чувствительности точность ухудшается в результате увеличения ложных срабатываний.
Среди производные сигналы, используемые для прогнозирования, представляют собой статистику, полученную на основе статистики подпоследовательности, такой как k-mer статистика, Isochore (генетика) или Compositional domain GC состав / однородность / энтропия, последовательность и длина кадра, интрон / экзон / донор / акцептор / промотор и сайт связывания рибосомы словарь, фрактальное измерение, преобразование Фурье псевдо- кодированная ДНК, Z-кривая параметры и некоторые особенности прогона.
Было высказано предположение, что сигналы, отличные от тех, которые непосредственно обнаруживаются в последовательностях, могут улучшить предсказание гена. Например, сообщалось о роли вторичной структуры в идентификации регуляторных мотивов. Кроме того, было высказано предположение, что предсказание вторичной структуры РНК помогает предсказывать места соединения.
Искусственные нейронные сети - это вычислительные модели, которые превосходят машинное обучение и распознавание образов. Нейронные сети должны быть обучены с использованием примеров данных, прежде чем они смогут обобщать экспериментальные данные и протестированы с эталонными данными. Нейронные сети способны находить приблизительные решения проблем, которые сложно решить алгоритмически, при условии наличия достаточного количества обучающих данных. Применительно к предсказанию генов нейронные сети могут использоваться наряду с другими методами ab initio для предсказания или идентификации биологических особенностей, таких как сайты сплайсинга. Один из подходов предполагает использование скользящего окна, которое пересекает данные последовательности с перекрытием. Результатом в каждой позиции является оценка, основанная на том, считает ли сеть, что окно содержит сайт сращивания донора или сайт сращивания акцептора. Окна большего размера обеспечивают большую точность, но также требуют большей вычислительной мощности. Нейронная сеть является примером датчика сигнала, поскольку ее цель - идентифицировать функциональный сайт в геноме.
Программы, такие как Maker, комбинируют внешние и ab initio подходы путем сопоставления белков и EST данных с геномом для подтверждения предсказаний ab initio. Augustus, который может использоваться как часть конвейера Maker, также может включать подсказки в форме выравнивания EST или профилей белков для повышения точности предсказания гена.
Поскольку все геномы многих различных видов секвенированы, многообещающим направлением в текущих исследованиях по поиску генов является подход сравнительной геномики.
Это основано на том принципе, что силы естественного отбора заставляют гены и другие функциональные элементы претерпевать мутации медленнее, чем остальная часть генома, поскольку мутации в функциональных элементах с большей вероятностью негативно повлияет на организм, чем мутации где-либо еще. Таким образом, гены могут быть обнаружены путем сравнения геномов родственных видов для выявления этого эволюционного давления в пользу сохранения. Этот подход был впервые применен к геномам мыши и человека с использованием таких программ, как SLAM, SGP и TWINSCAN / N-SCAN и CONTRAST.
TWINSCAN исследовали только синтению человека и мыши на предмет ищите ортологичные гены. Такие программы, как N-SCAN и CONTRAST, позволяли включать выравнивания от нескольких организмов или, в случае N-SCAN, одного альтернативного организма от мишени. Использование нескольких информаторов может привести к значительному повышению точности.
КОНТРАСТ состоит из двух элементов. Первый - это меньший классификатор, идентифицирующий сайты сплайсинга доноров и сайты сплайсинга акцепторов, а также стартовые и стоп-кодоны. Второй элемент включает построение полной модели с использованием машинного обучения. Разделение проблемы на два означает, что меньшие целевые наборы данных могут использоваться для обучения классификаторов, и этот классификатор может работать независимо и обучаться с меньшими окнами. Полная модель может использовать независимый классификатор и не тратить время вычислений или сложность модели на переклассификацию границ интрон-экзон. В статье, в которой представлен CONTRAST, предлагается классифицировать их метод (и методы TWINSCAN и т. Д.) Как сборку гена de novo, использующую альтернативные геномы и идентифицирующую его как отличную от ab initio, которая использует целевые «информативные» геномы.
Сравнительный поиск генов также можно использовать для проецирования высококачественных аннотаций из одного генома в другой. Известные примеры включают Projector, GeneWise, GeneMapper и GeMoMa. Такие методы сейчас играют центральную роль в аннотации всех геномов.
Псевдогены являются близкими родственниками генов, имеющих очень высокую гомологию последовательностей, но неспособных кодировать один и тот же продукт белок. Когда-то они считались побочными продуктами секвенирования генов, но по мере раскрытия регуляторных ролей они сами по себе становятся прогностическими целями. В прогнозировании псевдогенов используются существующие методы подобия последовательностей и ab initio методы, а также добавляется дополнительная фильтрация и методы определения характеристик псевдогенов.
Методы подобия последовательностей могут быть настроены для предсказания псевдогенов с использованием дополнительной фильтрации для поиска кандидатов псевдогенов. Это может использовать обнаружение отключения, которое ищет бессмысленные мутации или мутации со сдвигом рамки, которые могут усечь или разрушить функциональную кодирующую последовательность. Кроме того, трансляция ДНК в белковые последовательности может быть более эффективной, чем просто прямая гомология ДНК.
Датчики содержимого можно фильтровать в соответствии с различиями в статистических свойствах между псевдогенами и генами, такими как уменьшение количества CpG-островков в псевдогенах, или различия в содержании GC между псевдогенами и их соседями. Сигнальные сенсоры также можно отточить до псевдогенов, ища отсутствие интронов или полиадениновых хвостов.
Метагеномика - это исследование генетического материала, извлеченного из окружающей среды, в результате чего получают информацию о последовательности из пула организмов. Прогнозирование генов полезно для сравнительной метагеномики.
Инструменты метагеномики также делятся на основные категории, использующие либо подходы схожести последовательностей (MEGAN4), либо методы ab initio (GLIMMER-MG).
Glimmer-MG является расширением GLIMMER, которое в основном основывается на ab initio подходе к поиску генов и с использованием обучающих наборов из родственных организмов. Стратегия прогнозирования дополняется классификацией и кластеризацией наборов данных генов до применения методов ab initio прогнозирования генов. Данные сгруппированы по видам. Этот метод классификации использует методы метагеномной филогенетической классификации. Примером программного обеспечения для этой цели является Phymm, использующий интерполированные модели Маркова, и PhymmBL, который интегрирует BLAST в процедуры классификации.
MEGAN4 использует подход подобия последовательностей, используя локальное выравнивание с базами данных известных последовательностей, но также пытается классифицировать с использованием дополнительной информации о функциональных ролях, биологических путях и ферментах. Как и при прогнозировании генов отдельного организма, подходы схожести последовательностей ограничены размером базы данных.
FragGeneScan и MetaGeneAnnotator - популярные программы прогнозирования генов, основанные на скрытой марковской модели. Эти предикторы учитывают ошибки секвенирования, частичные гены и работают для коротких чтений.
Еще один быстрый и точный инструмент для прогнозирования генов в метагеномах - MetaGeneMark. Этот инструмент используется Объединенным институтом генома Министерства энергетики США для аннотирования IMG / M, самой большой коллекции метагеномов на сегодняшний день.