 | |
|---|---|
| Содержимое | |
| Описание | Полиморфизм одиночных нуклеотидов База данных |
| Организмы | Homo sapiens |
| Связаться | |
| Исследовательский центр | Национальный центр биотехнологической информации |
| Первичное цитирование | PMID 21097890 |
| Дата выпуска | 1998 |
| Доступ | |
| Формат данных | ASN.1, Fasta, XML |
| Веб-сайт | www.ncbi.nlm.nih.gov / snp / |
| URL загрузки | ftp://ftp.ncbi.nih.gov/snp/ |
| Веб-сервис URL | EUtils. SOAP |
База данных полиморфизма одиночных нуклеотидов (dbSNP) - это бесплатный общедоступный архив генетических вариаций внутри и между различными видами, разработанный и размещенный Национальный центр биотехнологической информации (NCBI) в сотрудничестве с Национальным исследовательским институтом генома человека (NHGRI). Хотя название базы данных подразумевает совокупность только одного класса полиморфизмов (т. Е. однонуклеотидных полиморфизмов (SNP)), на самом деле она содержит ряд молекулярных вариаций: (1) SNP, (2) короткие делеционные и инсерционные полиморфизмы (indels / DIPs), (3) микросателлитные маркеры или короткие тандемные повторы (STR), (4) полинуклеотидные полиморфизмы (MNP), (5) гетерозиготные последовательности и (6) названные варианты. DbSNP принимает очевидно нейтральные полиморфизмы, полиморфизмы, соответствующие известным фенотипам, и области без вариаций. Он был создан в сентябре 1998 года для дополнения GenBank, коллекции NCBI общедоступных последовательностей нуклеиновых кислот и белков.
В 2017 году NCBI прекратил поддержку всех нечеловеческих организмов в dbSNP. На момент сборки 153 (выпущенной в августе 2019 г.) dbSNP собрал почти 2 миллиарда представлений, представляющих более 675 миллионов различных вариантов для Homo sapiens.
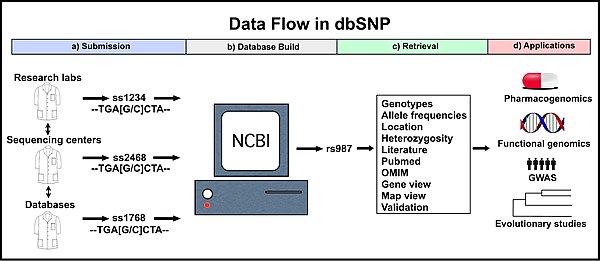 a) Различные источники предоставляют данные, и каждому варианту присваивается уникальный представленный SNP номер ID (ss #). б) dbSNP компилирует идентичные записи ss # в один эталонный кластер SNP (rs #), содержащий данные из каждого ss #. c) Пользователи могут извлекать данные для определенных записей rs # и анализировать эти вариации. г) Данные dbSNP помогают в клинических и прикладных исследованиях. Идентификаторы ss # и rs # на этом рисунке приведены только в качестве примера. NCBI, Национальный центр биотехнологической информации; OMIM, Интернет-Менделирующее наследование в человеке; GWAS, полногеномное исследование ассоциации.
a) Различные источники предоставляют данные, и каждому варианту присваивается уникальный представленный SNP номер ID (ss #). б) dbSNP компилирует идентичные записи ss # в один эталонный кластер SNP (rs #), содержащий данные из каждого ss #. c) Пользователи могут извлекать данные для определенных записей rs # и анализировать эти вариации. г) Данные dbSNP помогают в клинических и прикладных исследованиях. Идентификаторы ss # и rs # на этом рисунке приведены только в качестве примера. NCBI, Национальный центр биотехнологической информации; OMIM, Интернет-Менделирующее наследование в человеке; GWAS, полногеномное исследование ассоциации. dbSNP - это онлайн-ресурс, созданный для помощи исследователям биологии. Его цель - действовать в качестве единой базы данных, содержащей все идентифицированные генетические вариации, которые можно использовать для исследования широкого спектра генетически обусловленных природных явлений. В частности, доступ к молекулярным вариациям, каталогизированным в dbSNP, помогает фундаментальным исследованиям, таким как физическое картирование, популяционная генетика, исследования эволюционных взаимосвязей, а также возможность быстро и легко количественно оценить количество вариаций на данном участке представляет интерес. Кроме того, dbSNP направляет прикладные исследования в области фармакогеномики и ассоциации генетической изменчивости с фенотипическими признаками. Согласно веб-сайту NCBI, «Долгосрочные инвестиции в такие новые и захватывающие исследования [dbSNP] обещают не только продвинуть вперед биологию человека, но и произвести революцию в практике современной медицины».
Первоначально dbSNP принимает заявки на любой организм из самых разных источников, включая отдельные исследовательские лаборатории, совместные усилия по обнаружению полиморфизма, крупномасштабные центры секвенирования генома, другие базы данных SNP (например, SNP консорциум, HapMap и т. д.) и частные предприятия. 1 сентября 2017 года dbSNP прекратил принимать данные о вариантах, отличных от человека, а два месяца спустя его интерактивные веб-сайты и соответствующие службы NCBI перестали предоставлять данные о вариантах, отличных от человека. Теперь dbSNP принимает и представляет только данные о человеческих вариантах.
Каждый представленный вариант получает представленный идентификационный номер SNP («ss #»). Этот регистрационный номер является стабильным и уникальным идентификатором для данной заявки. Уникальные отправленные записи SNP также получают ссылочный номер идентификатора SNP («rs #»; «refSNP cluster»). Однако, скорее всего, в dbSNP будет отправлено более одной записи об изменении, особенно для клинически значимых вариантов. Чтобы приспособиться к этому, dbSNP обычно собирает идентичные представленные записи SNP в единую ссылочную запись SNP, которая также является уникальным и стабильным идентификатором (см. Ниже).
Чтобы отправить варианты в dbSNP, нужно сначала получить дескриптор отправителя, который идентифицирует лабораторию, ответственную за отправку. Затем автор должен заполнить файл для отправки, содержащий соответствующую информацию и данные. Отправленные записи должны содержать десять основных элементов информации, перечисленных в следующей таблице. Другая информация, необходимая для подачи, включает контактную информацию, информацию о публикации (название, журнал, авторов, год), тип молекулы (геномная ДНК, кДНК, митохондриальная ДНК, хлоропласт ДНК) и организм. Более подробную информацию о том, как отправить в dbSNP, можно найти по адресу: Как отправить в dbSNP
| Элемент | Объяснение |
|---|---|
| Контекст последовательности (обязательно) | Важный компонент отправки в dbSNP - это однозначное местоположение отправляемой вариации. dbSNP теперь минимально требует, чтобы вы отправляли вариант местоположения в качестве утвержденной позиции в последовательностях RefSeq или INSDC. |
| Аллели (обязательно) | Аллели определяют каждый класс вариации. dbSNP определяет однонуклеотидные варианты в своей схеме представления как G, A, T или C и не допускает неоднозначных кодов IUPAC, таких как N, в определении аллеля вариации. |
| Метод (обязательно) | Каждый отправитель определяет методы в своей заявке либо как методы, используемые для анализа вариаций, либо как методы, используемые для оценки частот аллелей. dbSNP группирует методы по классам методов для облегчения запросов с использованием общей экспериментальной техники в качестве поля запроса. Отправитель предоставляет все остальные сведения о методах в текстовом описании метода. |
| Утвержденное происхождение аллеля (обязательно) | Заявитель может предоставить заявление (утверждение) с подтверждающими экспериментальными доказательствами того, что вариант имеет конкретное аллельное происхождение. Утверждения для одного refSNP суммируются и получают значение атрибута зародышевой линии или неизвестно. |
| Популяция (обязательно) | Каждый отправитель определяет выборку популяции либо как группу, используемую для первоначального определения вариаций, либо как группу, используемую для определения специфичных для популяции измерений частот аллелей. Эти популяции могут быть одними и теми же в некоторых экспериментальных проектах. |
| Размер выборки (необязательно) | В dbSNP есть два поля размера выборки. В одном поле, SNPASSAY SAMPLE SIZE, указано количество хромосом в образце, использованное для первоначального установления или обнаружения вариации. Другое поле размера выборки, SNPPOPUSE SAMPLE SIZE, сообщает количество хромосом, используемых в качестве знаменателя при вычислении оценок частот аллелей. |
| Частоты аллелей для конкретных популяций (необязательно) | Данные о частотах отправляются в dbSNP в виде подсчета аллелей или интервалов сгруппированных частот, в зависимости от точности экспериментального метода, использованного для измерения. dbSNP содержит записи частот аллелей для конкретных выборок населения, которые определяются каждым отправителем и используются для проверки представленных вариантов. |
| Частоты генотипов для конкретных популяций (необязательно) | Подобно аллелям, генотипы имеют частоты в популяциях, которые могут быть отправлены в dbSNP, и используются для проверки представленных вариантов. |
| Индивидуальные генотипы | dbSNP принимает индивидуальные генотипы из образцов, предоставленных донорами, которые согласились на размещение их последовательности ДНК в общедоступной базе данных (например, HapMap или проект 1000 Genomes). |
| Информация о валидации (необязательно) | Анализы, подтвержденные непосредственно отправителем в разделе ВАЛИДАЦИЯ, показывают тип свидетельства, использованного для подтверждения вариации. |
Новая информация, полученная dbSNP, периодически становится доступной общественности в виде серии «сборок» (то есть версий и выпусков данных). Нет графика выпуска новых сборок; вместо этого сборки обычно выпускаются, когда становится доступной новая сборка генома, при условии, что с геномом связаны какие-то каталогизированные вариации. Это происходит примерно каждые 3–4 месяца. Последовательности генома могут быть улучшены с течением времени, поэтому эталонные SNP («refSNP») из предыдущих сборок, а также новые представленные SNP повторно отображаются на вновь доступную последовательность генома. Несколько отправленных SNP, если они сопоставлены с одним и тем же местоположением, группируются в один кластер refSNP, и им присваивается ссылочный идентификационный номер SNP. Однако, если обнаруживается, что две записи кластера refSNP сопоставлены с одним и тем же расположением (т. Е. Идентичны), dbSNP также объединит эти записи. В этом случае меньший идентификатор номера refSNP (т. Е. Самая ранняя запись) теперь будет представлять обе записи, а идентификаторы большего номера refSNP станут устаревшими. Эти устаревшие идентификаторы номеров refSNP больше не используются для новых записей. Когда происходит слияние двух записей refSNP, изменение отслеживается, и прежние идентификаторы номеров refSNP все еще могут использоваться в качестве поискового запроса. Этот процесс слияния идентичных записей уменьшает избыточность в dbSNP.
Есть два исключения из вышеуказанных критериев слияния. Во-первых, вариации разных классов (например, SNP и DIP) не объединяются. Во-вторых, клинически важные refSNP, которые цитировались в литературе, называются «драгоценными»; слияние, которое устранило бы такой refSNP, никогда не выполняется, поскольку в дальнейшем это может вызвать путаницу.
dbSNP можно найти с помощью поискового инструмента Entrez SNP. Для поиска можно использовать различные запросы: идентификатор номера ss, идентификатор номера refSNP, имя гена, экспериментальный метод, класс популяции, детализация популяции, публикация, маркер, аллель, хромосома, основание. положение, диапазон гетерозиготности или номер сборки. Кроме того, с помощью пакетных запросов можно получить одновременно множество результатов. Поиск возвращает идентификаторы номеров refSNP, которые соответствуют термину запроса, и сводку доступной информации для этого кластера refSNP.
Информация, доступная для кластера refSNP, включает в себя основную информацию из каждого отдельного представления (см. «Представление»), а также информацию, доступную путем объединения данных из нескольких представлений (например, гетерозиготность, частота генотипов). Для более глубокого изучения кластера refSNP доступно множество инструментов. Карта показывает положение вариации в геноме и других близлежащих вариациях. Другой инструмент, просмотр генов, сообщает о местоположении вариации в гене (если она есть в гене), о старом и новом кодоне, о аминокислотах, кодируемых обоими, и о том, является ли изменение синонимом или несинонимом. Программа просмотра последовательностей показывает положение варианта по отношению к интронам, экзонам и другим отдаленным и близким вариантам. Также доступно трехмерное картирование структуры, которое показывает трехмерные изображения кодируемого белка.
dbSNP также связан со многими другими ресурсами NCBI, включая нуклеотид, белок, ген, таксономию и структуры баз данных, а также PubMed, UniSTS, PMC, OMIM и UniGene.
В статусе проверки перечислены категории свидетельств, которые поддерживают вариант. К ним относятся: (1) несколько независимых представлений; (2) данные о частоте или генотипе; (3) подтверждение отправителя; (4) наблюдение всех аллелей по крайней мере в двух хромосомах; (5) генотипирован HapMap ; и (6) секвенировано в рамках проекта 1000 Genomes Project.
Качество данных, обнаруженных по dbSNP, подвергалось сомнению многими исследовательскими группами, которые подозревают высокий ложноположительный ставки из-за генотипирования и ошибок определения базы. Эти ошибки могут быть легко внесены в dbSNP, если отправитель использует (1) некритические биоинформатические сопоставления очень похожих, но различных последовательностей ДНК и / или (2) ПЦР с праймерами., которые не могут отличить похожие, но разные последовательности ДНК. Mitchell et al. (2004) проанализировали четыре исследования и пришли к выводу, что dbSNP имеет частоту ложноположительных результатов от 15 до 17% для SNP, а также что частота минорного аллеля превышает 10% примерно для 80% SNP, которые являются не ложные срабатывания. Аналогичным образом Musemeci et al. (2010) заявляет, что до 8,32% двуаллельных кодирующих SNP в dbSNP являются артефактами очень похожих последовательностей ДНК (то есть паралогичных генов) и называют эти записи однонуклеотидными различиями (SND). Высокий уровень ошибок в dbSNP не может вызывать удивления: из 23,7 миллиона записей refSNP для людей только 14,5 миллиона были подтверждены, а остальные 9,2 миллиона остались в качестве SNP-кандидатов. Однако, согласно Musemeci et al. (2010), даже код проверки, представленный в записи refSNP, полезен лишь частично: только проверка HapMap уменьшила количество SND (3% против 8%), но только принятие этого метода удаляет более половины реальных SNP в dbSNP. Эти авторы также отмечают, что один источник представлений от группы Ли страдает ошибками: 20% этих представлений являются SND (против 8% для представлений). Однако, как отмечают авторы, игнорирование всех этих представлений приведет к удалению многих реальных SNP.
Ошибки в dbSNP могут препятствовать исследованиям ассоциации генов-кандидатов и исследованиям на основе гаплотипа. Ошибки также могут увеличивать количество ложных выводов в ассоциативных исследованиях: увеличение количества SNP, которые проверяются путем проверки ложных SNP, требует дополнительных проверок гипотез. Однако эти ложные SNP на самом деле не могут быть связаны с признаками, поэтому альфа-уровень снижается больше, чем это необходимо для строгого теста, если были проверены только истинные SNP, а количество ложноотрицательных результатов увеличилось. Musemeci et al. (2010) предложили авторам исследований отрицательных ассоциаций проверить свои предыдущие исследования на предмет ложных SNP (SND), которые можно было бы удалить из анализа.
На отдельные последовательности можно ссылаться по их номерам идентификаторов кластеров refSNP (например, rs206437). На dbSNP следует ссылаться, используя 2001 Sherry et al. статья: Шерри, С.Т., Уорд, М.Х., Холодов, М., Бейкер, Дж., Фан, Л., Смигельски, Е.М., Сироткин, К. (2001). dbSNP: база данных генетических вариаций NCBI. Nucleic Acids Research, 29: 308-311.