 Двойная спираль ДНК
Двойная спираль ДНК Искусственный синтез гена или синтез гена, относится к группе методов, которые используются в синтетической биологии для конструирования и сборки генов из нуклеотидов de novo. В отличие от синтеза ДНК в живых, для искусственного синтеза генов требуется матричная ДНК, которая позволяет синтезировать практически любую последовательность ДНК в лаборатории. Он состоит из двух основных этапов, первый из которых - твердофазный синтез ДНК, иногда известный как печать ДНК. В результате образуются олигонуклеотидные фрагменты, которые обычно составляют менее 200 пар оснований. Затем второй этап включает соединение этих олигонуклеотидных фрагментов с использованием различных методов сборки ДНК. Не требуется теоретически полностью синтетические молекулы ДНК без ограничений по нуклеотидной ДНК.
Синтез первого полного гена, дрожжевой тРНК, былан Хар Гобинд Хораной и сотрудниками в 1972 году. Синтез первого пептида - и гены, кодирующие белок, были выполнены в лабораториях Герберта Бойера и Александра Маркхэма соответственно. Совсем недавно были разработаны методы искусственного синтеза генов, которые позволяют собирать целые хромосомы и геномы. Первая синтетическая дрожжевая хромосома синтезирована в 2014 году, а также были синтезированы целые функциональные бактериальные хромосомы. Кроме того, в искусственном синтезе генов в творческой семье новые пары азотистых оснований (неестественные пары оснований).
Олигонуклеотиды химически синтезируются с использованием строительных блоков, называемых нуклеозидными фосфорамидитами. Это могут быть нормальные или модифицированные нуклеозиды, которые имеют защитные группы для предотвращения неправильного взаимодействия их аминов, гидроксильных и фосфатных групп. За один раз увеличить один фосфорамидит, снять защиту с 5-гидроксильной группы, добавить новое основание и так далее. Цепь растет в направлении от 3 'до 5', что является обратным по отношению к биосинтезу. В конце все защитные группы удаляются. Это не менее, поскольку это химический процесс, происходит несколько неправильных взаимодействий, приводящих к появлению дефектных продуктов. Чем длиннее синтезируемая олигонуклеотидная последовательность, тем больше дефектов, поэтому этот процесс применим только для получения коротких последовательностей нуклеотидов. Текущий практический предел составляет около 200 п.н. (пар оснований ) для олигонуклеотида с достаточным качеством для непосредственного использования в биологических целях. ВЭЖХ можно использовать для выделения продуктов с правильной последовательностью. Между тем, большое количество олигонуклеотидов может быть синтезировано на параллельно генных чипах. Для оптимального выполнения процедур следует их готовить индивидуально и в больших масштабах.
Обычно набором индивидуально разработанных олигонуклеотидов изготавливается на автоматических твердофазных синтезаторах, очищается и соединяется посредством специального отжига и стандартного реакции лигирования или полимеразы. Для повышения специфичности отжига олигонуклеотидов на стадии синтеза используется набор термостабильных ферментов ДНК лигаза и полимераза . На сегодняшний день описано несколько методов алгоритмов генов, таких как лигирование фосфорилированных перекрывающихся олигонуклеотидов, метод Fok I и модифицированная форма лигазной цепной реакции для синтеза гена. Кроме того, описано несколько подходов к сборке ПЦР. Обычно в них используются олигонуклеотиды длиной 40-50 нуклеотидов, которые перекрывают друг друга. Эти олигонуклеотиды предназначены для покрытия большей части своей цепей, и полноразмерная молекула постепенно генерируется с помощью ПЦР с удлинением с перекрытием (OE), термодинамически сбалансированной ПЦР наизнанку (TBIO) или комбинированных подходов. Чаще всего синтезируемые фрагменты имеют размер от 600 до 1200 п.н., хотя намного более длинные гены были получены ранее собранных фрагментов размером менее 1000 п.н. В этом диапазоне размеров необходимо протестировать несколько клонов-кандидатов, подтверждающих последовательность клонированного синтетического гена с помощью методов автоматического секвенирования.
Более того, поскольку сбор продукта полноразмерного гена зависит от эффективного и специфического выравнивания одноцепочечных олигонуклеотидов, критические параметры для успеха включают расширенной состав, содержащие вторичные структуры, вызванные инвертированными повторами, самым высоким или низкий уровень GC или повторяющиеся структуры. Обычно эти сегменты конкретного гена можно синтезировать путем разделения процедур нескольких последовательных шагов и окончательной сборки более коротких подпоследовательностей, что, в свою очередь, приводит к значительному увеличению времени и трудозатрат, необходимое для его получения. Результат эксперимента по синтезу генов сильно зависит от качества используемых олигонуклеотидов. Для этих протоколов синтеза генов на отжига продукта прямо и экспоненциально зависит от правильности используемого олигонуклеотидов. В качестве альтернативы после выполнения синтеза глоссарий с олигонуклеотидами более низкого качества необходимо приложить больше усилий для последующего качества во время анализа клонов, который обычно выполняется с помощью длительных стандартных процедур клонирования и секвенирования. Другая проблема, связанная со всеми современными методами синтеза генов, высокая частота ошибок из-за использования химически синтезированных олигонуклеотидов. Частота использования увеличенного размера увеличивающегося количества олигонуклеотидов, как следствие правильного продукта, увеличивается по мере увеличения количества олигонуклеотидов. Проблема мутации может быть решена с помощью более коротких олигонуклеотидов, используемых для сборки гена. Однако все методы сборки на основе отжига требуют, чтобы праймеры были смешаны вместе в одной пробирке. В этом случае более короткие перекрытия не всегда позволяют точный и специфический отжиг комплементарных праймеров, что приводит к ингибированию полноразмерных продуктов. Ручной дизайн олигонуклеотидов - трудоемкая процедура и не гарантирует успешного выполнения желаемого гена. Для оптимальной работы почти всех методов, основанных на отжиге, температурных перекрывающихся областях должны быть одинаковыми для всех олигонуклеотидов. Необходимая оптимизация праймера с использованием программного обеспечения дизайна олигонуклеотидов. Настоящее время было представлено несколько решений для автоматизированного конструирования праймеров для синтеза гена.
Для преодоления проблем, связанных с качеством олигонуклеотидов, было разработано несколько сложных схем с использованием либо отдельно полученных рыболовных олигонуклеотидов, ферменты, связывающие несовпадение, семейство mutS или специфические эндонуклеазы из бактерий или фагов. Тем не менее, все эти стратегии увеличивают время и затраты на синтез генов, основанный на отжиге химически синтезированных олигонуклеотидов.
Массивно параллельное секвенирование также использовалось в качестве инструмента для скрининга сложных библиотек олигонуклеотидов и возможности поиска точных молекул. В одном подходе олигонуклеотиды секвенируются на платформе пиросеквенирования 454, и роботизированная система изображения и выбирает отдельные шарики, соответствующие точной последовательности. Другим подходом сложная библиотека олигонуклеотидов модифицируется уникальными фланкирующими тегами перед массовым параллельным секвенированием. Затем праймеры, ориентированные на метки, позволяют извлекать молекулы с желаемыми последовательностями с помощью выбора ПЦР.
Все чаще гены упорядочиваются в наборах, включая функционально связанные гены или несколько вариантов последовательностей одного гена. Практически все разрабатываемые терапевтические белки, такие как моноклональные антитела, оптимизированы путем многих вариантов генов на улучшенную функцию или экспрессию.
В то время как традиционные нуклеиновые кислоты используют только 4 пары оснований - аденин, тимин, гуанин и цитозин, синтез олигонуклеотидов в будущем использование неприродных пар оснований, которые являются искусственно созданные и синтезированные азотистые основания, не встречающиеся в природе.
В 2012 году группа ученых во главе с Флойдом Ромесбергом, химическим биологом из Исследовательского института Скриппса в Сан-Диего, Калифорния, опубликовала, что его команда разработала неестественную пару оснований (UBP). Два новых искусственных нуклеотида или пара неестественных оснований (UBP) были названы d5SICS и dNaM. С технической точки зрения, эти искусственные нуклеотиды, несущие гидрофобные азотистые основания, содержат два слитых ароматических кольца, которые образуют комплекс (d5SICS - dNaM) или пару оснований в ДНК. В 2014 году та же команда из Исследовательского института Скриппса сообщила, что они синтезируют отрезок кольцевой ДНК, известную как плазмида, обеспечивают естественные пары оснований TA и CG, а также наиболее эффективную лабораторию UBP, созданную Ромесбергом. его в клеточной среде, которая успешно реплицировала неестественные пары оснований в течение нескольких поколений. Это первый известный пример передачи живым организмом расширенного генетического кода последующим поколениям. Частично это было достигнуто путем добавления поддерживающего гена водорослей, который экспрессирует переносчик нуклеотидтрифосфата, который эффективно импортирует трифосфаты как d5SICSTP, так и dNaMTP в бактерии E. coli. Затем естественные пути репликации бактерий используют их для точной репликации плазмиды, компонент d5SICS-dNaM.
Успешное включение третьей пары является значительным прорывом в достижении цели значительного увеличения аминокислот, которые могут кодироваться ДНК, используя 20 аминокислот до теоретически возможно 172, тем самым расширяя возможности живых организмов выполнить новые белки. В эти неестественные пары оснований можно будет синтезировать и в будущем включить олигонуклеотиды с помощью методов печати ДНК.
Система ДНК, таким образом, система передачи ДНК, которая определяет как ДНК, кодирующие конкретную биологическую функцию (например, промоторы, регуляторные последовательности) транскрипции или открытые рамки считывания ). Однако, поскольку синтез олигонуклеотидов обычно не может точно выполнять олигонуклеотидов длиной более нескольких сотен пар оснований, необходимо использовать методы сборки ДНК этих частей вместе для создания функциональных генов, мультигенных цепей или даже целых синтетических хромосом или геномов. Некоторые методы сборки ДНК определяют только протоколы частей ДНК, в то время как другие методы также определяют правила для форм частей ДНК, которые с ними совместимы. Эти процессы можно масштабировать, чтобы обеспечить сборку целых хромосом или геномов. В 2015 году было разработано 14 различных стандартов сборки, каждый со своими плюсами и минусами. В процессе разработки стандартов сборки ДНК упростила рабочий процесс синтетической биологии, помогла создать модульные системы сборки ДНК.
Различные методы сборки ДНК могут быть разделены на три основных категории - сборка, опосредованная эндонуклеазами, сайт-специфическая рекомбинация и сборка на основе длинного перекрытия. Группа методов имеет свои отличительные особенности, а также свои преимущества и каждую ограничение.
Эндонуклеазы - это ферменты, которые распознают и расщепляют сегменты нуклеиновых кислот, и их можно использовать для управления сборкой ДНК. Из различных типов рестрикционных ферментов рестрикционные ферменты типа II являются наиболее общедоступными и используются, поскольку их сайты расщепления расположены рядом или в их сайтах узнавания. Следовательно, методы сборки, опосредованные эндонуклеазами, используют это свойство для частей ДНК и протоколов сборки.
 BBF RFC 10 сборка из двух частей, совместимых с BioBricks. Обработка вышестоящего фрагмента EcoRI и SpeI, нижележащего фрагмента EcoRI и XbaI обеспечивает сборку в желаемую последовательность. SpeI и XbaI образуют дополнительные выступы вместе, они соединяют два фрагмента ДНК вместе. Все исходные сайты рестрикции сохранятся в конечной конструкции, которую можно использовать для дальнейших исходных вариантов BioBricks.
BBF RFC 10 сборка из двух частей, совместимых с BioBricks. Обработка вышестоящего фрагмента EcoRI и SpeI, нижележащего фрагмента EcoRI и XbaI обеспечивает сборку в желаемую последовательность. SpeI и XbaI образуют дополнительные выступы вместе, они соединяют два фрагмента ДНК вместе. Все исходные сайты рестрикции сохранятся в конечной конструкции, которую можно использовать для дальнейших исходных вариантов BioBricks. Стандарт сборки BioBricks был описан и введен Томом Найтом в 2003 году и с тех пор постоянно обновляется. В настоящее время наиболее часто используемым стандартом BioBricks является стандартной сборкой 10 или BBF RFC 10. BioBricks обеспечивает последовательность префиксов и суффиксов, необходимых для совместимости части ДНК с помощью метода сборки BioBricks, позволяя объединить вместе все части ДНК, которые имеют формат BioBricks.
Префикс содержит сайты рестрикции для EcoRI, NotI и XBaI, а суффикс содержит сайты рестрикции SpeI, NotI и PstI. Вне области префикса и суффикса часть ДНК не должна содержать эти сайты рестрикции. Чтобы соединить две части BioBrick вместе, одна из плазмид расщепляется EcoRI и SpeI, а вторая плазмида расщепляется EcoRI и XbaI. Два выступа EcoRI дополнительные выступы друг и таким образом отжигаются вместе, в то время как SpeI и XbaI также образуют дополнительные выступы, которые также можно лигировать вместе. Указанная плазмида показывает исходные префикса и суффикса, ее можно использовать для соединения с другими частями BioBricks. Из-за этого стандарта сборки BioBricks считается идемпотентным по своей природе. Однако между двумя слитыми BioBricks будет также образовываться«рубцовая» последовательность (TACTAG или TACTAGAG). Это предотвращает использование BioBricks для создания слитых белков, последовательность из 6 пар оснований кодирует тирозин и стоп-кодон, что приводит к прекращению трансляции после экспрессии первого домена, в то время как последовательность длиной 8 пар оснований вызывает сдвиг рамки , предотвращает постоянное считывание кодонов. Чтобы предложить альтернативные рубцовые наборы, которые, например, дают рубцы, не содержащие стоп-кодонов, были разработаны другие стандарты сборки, такие как BB-2 Assembly, BglBricks Assembly, Silver Assembly и Freiburg Assembly.
Хотя вышеупомянутый самый простой метод сборки деталей описан BioBrick, используется несколько других широко используемых методов сборки, которые обеспечивают несколько преимуществ по сравнению со стандартной сборкой. Сборка из 3 антибиотиков (3A) позволяет выбрать правильную сборку путем выбора антибиотика, в то время как сборка амплифицированной вставки преодолевает низкую эффективность трансформации, наблюдаемую при сборке 3A.
Стандарт сборки BioBrick также служил в качестве вдохновения для использования других типов эндонуклеаз для сборки ДНК. Например, как стандарт iBrick, так и стандарты сборки приложения HomeRun используют эндонуклеазы самонаведения вместо рестрикционных ферментов типа II.
Некоторые методы сборки также используют рестрикцию типа II. эндонуклеазы. Они отличаются от других эндонуклеаз типа II, поскольку они отрезают несколько пар оснований от сайта узнавания. В результате последовательность выступа может быть изменена, чтобы содержать желаемую последовательность. Это обеспечивает методы сборки Типа II с двумя преимуществами: он обеспечивает сборку без образования рубцов и позволяет производить сборку из нескольких частей в одной ванне. Методы сборки, в которых используются эндонуклеазы типа II, включают Golden Gate и связанные с ним варианты.
 Последовательность частей ДНК для сборки Golden Gate может быть направлена путем определения уникальных дополнительных выступов для каждой части. Таким образом, чтобы собрать ген 1 в порядке фрагментов A, B и C, 3'-выступ для фрагмента A комплементарен 5'-выступу для фрагмента B, и аналогично для фрагмента B и фрагмента C. Для целевой плазмиды выбираемый Маркер фланкирован сайтами рестрикции BsaI, направленными наружу. Это иссекает селектируемый маркер, позволяя вставить окончательную конструкцию. Лигаза Т4 используется для лигирования фрагментов вместе и с целевой плазмидой.
Последовательность частей ДНК для сборки Golden Gate может быть направлена путем определения уникальных дополнительных выступов для каждой части. Таким образом, чтобы собрать ген 1 в порядке фрагментов A, B и C, 3'-выступ для фрагмента A комплементарен 5'-выступу для фрагмента B, и аналогично для фрагмента B и фрагмента C. Для целевой плазмиды выбираемый Маркер фланкирован сайтами рестрикции BsaI, направленными наружу. Это иссекает селектируемый маркер, позволяя вставить окончательную конструкцию. Лигаза Т4 используется для лигирования фрагментов вместе и с целевой плазмидой. Протокол сборки Golden Gate был определен Engler et al. 2008, чтобы определить метод сборки ДНК, который дал бы окончательную конструкцию без рубцовой последовательности, а также без исходных сайтов рестрикции. Это позволяет белку экспрессироваться без содержания нежелательных белковых последовательностей, которые могут отрицательно повлиять на укладку или экспрессию белка. При использовании рестрикционного фермента BsaI, который производит выступающую часть из 4 пар оснований, для сборки можно использовать до 240 уникальных непалиндромных последовательностей.
Конструирование и сборка плазмиды
При клонировании Golden Gate каждый фрагмент ДНК должен быть собран помещен в плазмиду, фланкированную обращенными внутрь сайтами рестрикции BsaI, содержащими запрограммированные выступающие последовательности. Для каждого фрагмента ДНК 3'-выступающая последовательность комплементарна 5'-выступающей части следующего нижележащего фрагмента ДНК. Для первого фрагмента 5'-выступ комплементарен 5'-выступу целевой плазмиды, в то время как 3'-выступ последнего фрагмента комплементарен 3'-выступу целевой плазмиды. Такая конструкция позволяет собрать все фрагменты ДНК в реакции в одном сосуде (где все реагенты смешиваются вместе), при этом все фрагменты расположены в правильной последовательности. Успешно собранные конструкции отбираются путем обнаружения потери функции кассеты для скрининга, которая была первоначально в целевой плазмиде.
MoClo и Golden Braid
Исходная сборка Golden Gate позволяет создать только одну конструкцию в целевой плазмиде. вектор. Чтобы позволить использовать эту конструкцию в последующей реакции в качестве вектора входа, были разработаны стандарты MoClo и Golden Braid.
Стандарт MoClo включает определение нескольких уровней сборки ДНК:
 Стандарт сборки MoClo позволяет для конструкций Golden Gate, которые будут собираться на последующих уровнях. В приведенном здесь примере четыре гена, собранные с помощью сборки Golden Gate 1 уровня, собираются в мультигенную конструкцию в сборке уровня 2.
Стандарт сборки MoClo позволяет для конструкций Golden Gate, которые будут собираться на последующих уровнях. В приведенном здесь примере четыре гена, собранные с помощью сборки Golden Gate 1 уровня, собираются в мультигенную конструкцию в сборке уровня 2. 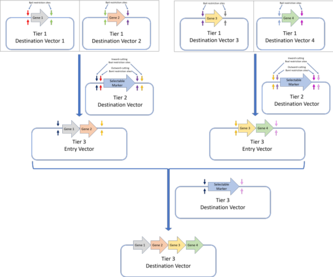 Стандарт сборки Golden Braid также строится на первом уровне сборки Golden Gate и собирает последующие уровни по парному протоколу. Четыре вектора назначения уровня 1 (собранные с помощью сборки Golden Gate) собираются в два вектора назначения уровня 2, которые затем используются в качестве векторов входа
Стандарт сборки Golden Braid также строится на первом уровне сборки Golden Gate и собирает последующие уровни по парному протоколу. Четыре вектора назначения уровня 1 (собранные с помощью сборки Golden Gate) собираются в два вектора назначения уровня 2, которые затем используются в качестве векторов входа