Нуклеоид (то есть ядро -подобное) представляет собой область неправильной формы в прокариотическую клетке, которая содержит все или большую часть генетического материала. Хромосома из прокариот является круговым, а его длина очень велики по сравнению с размерами ячеек, нуждающихся в его быть уплотнен, чтобы соответствовать. В отличие от ядра в виде эукариотической клетки, он не окружен ядерной мембраной. Вместо этого нуклеоид образуется путем конденсации и функционального расположения с помощью архитектурных белков хромосом и молекул РНК, а также суперспирализации ДНК. Длина генома варьируется в широких пределах (обычно не менее нескольких миллионов пар оснований), и клетка может содержать несколько его копий.
Структуры бактериального нуклеоида с высоким разрешением еще не известны, однако ключевые особенности были исследованы на Escherichia coli как на модельном организме. У E. coli хромосомная ДНК в среднем отрицательно свернута и свернута в плектонемические петли, которые ограничены разными физическими областями и редко диффундируют друг в друга. Эти петли пространственно организуются в области размером с мегабазы, называемые макродоменами, внутри которых участки ДНК часто взаимодействуют, но между которыми взаимодействия редки. Конденсированная и пространственно организованная ДНК образует спиральный эллипсоид, который радиально ограничен клеткой. Трехмерная структура ДНК в нуцеиде, по-видимому, варьируется в зависимости от условий и связана с экспрессией генов, так что архитектура нуклеоида и транскрипция гена тесно взаимосвязаны, взаимно влияя друг на друга.
 Формирование нуклеоида Escherichia coli A. Иллюстрация открытой конформации кольцевого генома Escherichia coli. Стрелки обозначают двунаправленную репликацию ДНК. Генетическая позиция начала репликации двунаправленного ДНК ( Oric) и участок хромосома decatenation ( DIF) в области терминации репликации ( тер) отмечена. Цвета представляют определенные сегменты ДНК, как описано в C. B. Иллюстрация случайной формы спирали, принятой чистой кольцевой ДНК Escherichia coli при тепловом равновесии без суперспиралей и дополнительных стабилизирующих факторов. C. Рисунок хромосомы только что родившейся клетки Escherichia coli. Геномная ДНК не только конденсирована в 1000 раз по сравнению с ее чистой случайной формой спирали, но также пространственно организована. oriC и dif локализованы в средней клетке, а определенные области ДНК, обозначенные цветами в A, организуются в пространственно различные домены. У E. coli идентифицировано шесть пространственных доменов. Четыре домена (Ori, Ter, Left и Right) структурированы, а два (NS-right и NS-left) неструктурированы. Конденсированная и организованная форма ДНК вместе со связанными с ней белками и РНК называется нуклеоидом.
Формирование нуклеоида Escherichia coli A. Иллюстрация открытой конформации кольцевого генома Escherichia coli. Стрелки обозначают двунаправленную репликацию ДНК. Генетическая позиция начала репликации двунаправленного ДНК ( Oric) и участок хромосома decatenation ( DIF) в области терминации репликации ( тер) отмечена. Цвета представляют определенные сегменты ДНК, как описано в C. B. Иллюстрация случайной формы спирали, принятой чистой кольцевой ДНК Escherichia coli при тепловом равновесии без суперспиралей и дополнительных стабилизирующих факторов. C. Рисунок хромосомы только что родившейся клетки Escherichia coli. Геномная ДНК не только конденсирована в 1000 раз по сравнению с ее чистой случайной формой спирали, но также пространственно организована. oriC и dif локализованы в средней клетке, а определенные области ДНК, обозначенные цветами в A, организуются в пространственно различные домены. У E. coli идентифицировано шесть пространственных доменов. Четыре домена (Ori, Ter, Left и Right) структурированы, а два (NS-right и NS-left) неструктурированы. Конденсированная и организованная форма ДНК вместе со связанными с ней белками и РНК называется нуклеоидом. У многих бактерий хромосома представляет собой одну ковалентно замкнутую (кольцевую) двухцепочечную молекулу ДНК, которая кодирует генетическую информацию в гаплоидной форме. Размер ДНК варьируется от 500 000 до нескольких миллионов пар оснований (п.н.), кодирующих от 500 до нескольких тысяч генов, в зависимости от организма. Хромосомная ДНК присутствует в клетках в очень компактной организованной форме, называемой нуклеоидом (что означает ядро-подобная), которая не заключена в ядерную мембрану, как в эукариотических клетках. Выделенный нуклеоид содержит 80% ДНК, 10% белка и 10% РНК по весу.
Грамотрицательные бактерии кишечной палочки является модельной системой для нуклеоидов исследований, как хромосомные ДНК становятся нуклеоидом, факторы участвуют в нем, что известно о его структуре, и как часть ДНК структурных аспекты влияют на экспрессию генов.
Есть два основных аспекта образования нуклеоидов; конденсация большой ДНК в маленькое клеточное пространство и функциональная организация ДНК в трехмерной форме. Гаплоидная кольцевая хромосома E. coli состоит из ~ 4,6 x 10 6 п.н. Если ДНК расслаблена в форме B, она будет иметь окружность ~ 1,5 миллиметра (0,332 нм x 4,6 x 10 6). Однако большая молекула ДНК, такая как хромосомная ДНК E. coli, не остается прямой жесткой молекулой в суспензии. Броуновское движение вызовет искривление и изгибы ДНК. Максимальная длина, до которой двойная спираль ДНК остается прямой, сопротивляясь изгибу, вызванному броуновским движением, составляет ~ 50 нм или 150 п.н., что называется персистентной длиной. Таким образом, чистая ДНК становится существенно конденсированной без каких-либо дополнительных факторов; при тепловом равновесии он принимает случайную форму спирали. Случайная спираль хромосомной ДНК E. coli будет занимать объем (4/3 π r 3) ~ 523 мкм 3, рассчитанный из радиуса вращения ( R g = (√N a) / √6), где a - Длина Куна (2 х персистентная длина), а N - количество сегментов длины Куна в ДНК (общая длина ДНК, деленная на а). Хотя ДНК уже сконденсирована в форме случайной спирали, она все еще не может принимать объем нуклеоида меньше микрона. Таким образом, присущего ДНК свойства недостаточно: дополнительные факторы должны способствовать дальнейшей конденсации ДНК порядка ~ 10 3 (объем случайной спирали, деленный на объем нуклеоида). Второй важный аспект образования нуклеоидов - это функциональное устройство ДНК. Хромосомная ДНК не только конденсирована, но и функционально организована способом, совместимым с процессами транзакций ДНК, такими как репликация, рекомбинация, сегрегация и транскрипция. Почти пять десятилетий исследований, начавшихся в 1971 году, показали, что окончательная форма нуклеоида возникает в результате иерархической организации ДНК. В самом маленьком масштабе (1 т.п.н. или меньше) нуклеоид-ассоциированные архитектурные белки ДНК конденсируются и организуют ДНК путем изгибания, образования петель, образования мостиков или обертывания ДНК. В более крупном масштабе (10 т.п.н. или больше) ДНК образует плектонемные петли, плетеную форму ДНК, индуцированную суперспирализацией. В масштабе мегабаз плектонемические петли сливаются в шесть пространственно организованных доменов (макродоменов), которые определяются более частыми физическими взаимодействиями между сайтами ДНК в одном и том же макродомене, чем между разными макродоменами. Длинные и короткие связи ДНК-ДНК, сформированные внутри и между макродоменами, способствуют конденсации и функциональной организации. Наконец, нуклеоид представляет собой спиральный эллипсоид с участками сильно конденсированной ДНК на продольной оси.
 Нуклеоид в масштабе ≥1 т.п.н. Организация ДНК нуклеоид-ассоциированными белками. ДНК изображена в виде серой прямой или изогнутой линии, а белки, связанные с нуклеоидами, изображены в виде синих сфер.
Нуклеоид в масштабе ≥1 т.п.н. Организация ДНК нуклеоид-ассоциированными белками. ДНК изображена в виде серой прямой или изогнутой линии, а белки, связанные с нуклеоидами, изображены в виде синих сфер. У эукариот геномная ДНК конденсируется в виде повторяющегося массива ДНК-белковых частиц, называемых нуклеосомами.
Нуклеосома состоит из ~ 146 п.н. ДНК, обернутой вокруг октамерного комплекса гистоновых белков. Хотя бактерии не имеют гистонов, они обладают группой ДНК-связывающих белков, называемых нуклеоид-ассоциированными белками (NAP), которые функционально аналогичны гистонам в широком смысле. NAP очень распространены и составляют значительную часть белкового компонента нуклеоида.
Отличительной характеристикой NAP является их способность связывать ДНК как специфическим (либо последовательностным, либо структурно-специфическим), так и неспецифическим для последовательности образом. В результате NAP являются белками с двойной функцией. Специфическое связывание NAP в основном участвует в ген-специфической транскрипции, репликации ДНК, рекомбинации и репарации. На пике их численности количество молекул многих NAP на несколько порядков превышает количество специфических сайтов связывания в геноме. Следовательно, считается, что NAP связываются с хромосомной ДНК в основном в режиме, не специфичном для последовательности, и именно этот режим является решающим для уплотнения хромосом. Примечательно, что так называемое связывание NAP, не специфичное для последовательности, может быть не полностью случайным. Может быть специфичность низкой последовательности и / или структурная специфичность из-за зависимой от последовательности конформации ДНК или конформации ДНК, созданной другими NAP.
Хотя молекулярные механизмы того, как NAP конденсируют ДНК in vivo, не совсем понятны, на основании обширных исследований in vitro выяснилось, что NAP участвуют в уплотнении хромосом посредством следующих механизмов: NAP индуцируют и стабилизируют изгибы в ДНК, тем самым способствуя конденсации ДНК за счет уменьшения продолжительность настойчивости. NAP конденсируют ДНК путем образования мостиков, обертывания и группирования, которые могут происходить между соседними сегментами ДНК или удаленными сегментами ДНК хромосомы. Другой механизм, с помощью которого NAP участвуют в уплотнении хромосом, заключается в ограничении отрицательных суперспиралей в ДНК, тем самым способствуя топологической организации хромосомы.
В E. coli идентифицировано не менее 12 NAP, наиболее изученными из которых являются HU, IHF, H-NS и Fis. Их количество и свойства связывания ДНК, а также влияние на конденсацию и организацию ДНК суммированы в таблицах ниже.
| Протеин | Молекулярная масса (кДа) | Штатный функциональный блок | Изобилие 1 в фазе роста | Обилие 1 в стационарной фазе |
|---|---|---|---|---|
| HUα и HUβ | ~ 9 | Гомо- и гетеродимеры | 55 000 (23) | 30 000 (12,5) |
| IHFα и IHFβ | ~ 11 | Гетеродимер | 12 000 (5) | 55 000 (23) |
| H-NS | ~ 15 | Гомодимер | 20 000 (8) | 15 000 (6) |
| Fis | ~ 11 | Гомодимер | 60 000 (25) | Необнаруживаемый |
| Дпс | ~ 19 | Додекамер | 6000 (0,4) | 180 000 (12,5) |
1 Данные по численности (молекулы / клетка) были взяты из; Число в скобках представляет собой микромолярную концентрацию, рассчитанную по следующей формуле: (количество нативных функциональных единиц / число Авогадро) x (1 / объем клетки в литре) x 10 3. Объем клеток в литрах (2 × 10 -15) определяли, принимая объем клетки E. coli равным 2 мкм 3.
| Протеин | Обязательный мотив | Аффинность специфического связывания ДНК 1 | Аффинность случайного связывания ДНК 1 |
|---|---|---|---|
| HU | Структурный мотив, определяемый изгибами и изгибами ДНК. | 7,5 х 10 −9 | 4,0 х 10 −7 |
| H-NS | WATCAANNNNTTR | 1,5 х 10 −9 | 1,7 х 10 −6 |
| IHF | TCGATAAATT | 10-15 х 10 -9 | 6 х 10 −8 |
| Fis | GNTYAAAWTTTRANC | 0,2-1,0 х 10 -9 | gt; 8,0 х 10 −6 |
| Дпс | ND | ND | 1,65 х 10 −7 |
| MatP | GTGACRNYGTCAC | 8,0 х 10 −9 | ND |
| MukBEF | ND | ND | ND |
1 Сродство связывания относится к константе равновесной диссоциации (Kd) в молярных единицах (M). ND = не определено
Гистоноподобный белок из штамма E. coli U93 (HU) является эволюционно консервативным белком бактерий. HU существует в E. coli в виде гомо- и гетеродимеров двух субъединиц HUα и HUβ, имеющих 69% аминокислотной идентичности. Хотя его называют гистоноподобным белком, близкими функциональными родственниками HU у эукариот являются белки группы высокой подвижности (HMG), а не гистоны. HU представляет собой ДНК-связывающий белок, не специфичный для последовательности. Он с низким сродством связывается с любой линейной ДНК. Однако он предпочтительно связывается с высоким сродством со структурно искаженной ДНК. Примеры искаженных субстратов ДНК включают в себя крестообразную ДНК, вдавленный ДНК, содержащие дц одноцепочечного перерыва, такие как ники, зазоры, или вилки. Кроме того, HU специфически связывает и стабилизирует опосредованную белком петлю ДНК. В структурно-специфическом режиме связывания ДНК HU распознает общий структурный мотив, определяемый изгибами или перегибами, созданными искажением, тогда как он связывается с линейной ДНК, блокируя фосфатный остов. В то время как с высоким сродством структурно-специфическое связывание требуется для специализированных функций, таких как HU сайт-специфической рекомбинации, репарации ДНК, репликации ДНК инициации и регуляции генов, по- видимому, что низкоаффинные общего связывания участвует в конденсации ДНК. При иммунопреципитации хроматина в сочетании с секвенированием ДНК ( ChIP-Seq ) HU не обнаруживает каких-либо специфических событий связывания. Вместо этого он демонстрирует равномерное связывание по всему геному, предположительно, отражая его в основном слабое, неспецифическое связывание, не связанное с последовательностью, таким образом маскируя связывание с высоким сродством in vivo.
В штаммах, лишенных HU, нуклеоид «деконденсируется», что согласуется с ролью HU в уплотнении ДНК. Следующие исследования in vitro предполагают возможные механизмы того, как HU может конденсироваться и организовывать ДНК in vivo. HU не только стабильно связывается с искаженной ДНК с изгибами, он вызывает гибкие изгибы даже в линейной ДНК при концентрации менее 100 нМ. Напротив, HU демонстрирует противоположный архитектурный эффект на ДНК при более высоких физиологически значимых концентрациях. Он образует жесткие нуклеопротеиновые нити, вызывающие растяжение ДНК, а не изгиб. Филаменты могут дополнительно образовывать сеть ДНК (группирование ДНК), расширяемую как латерально, так и медиально из-за мультимеризации HU-HU, запускаемой неспецифическим связыванием ДНК.
Как такое поведение HU актуально внутри клетки? Для образования филаментов требуется связывание HU с ДНК с высокой плотностью, один димер HU на 9-20 п.н. ДНК. Но существует только один димер HU на каждые ~ 150 п.н. хромосомной ДНК, исходя из предполагаемой численности димеров HU в 30 000 п.о. на клетку (4600000 п.н. / 30 000). Это указывает на то, что гибкие изгибы чаще возникают in vivo. Гибкий изгиб вызовет конденсацию из-за уменьшения длины сохранения ДНК, как показали эксперименты с магнитным пинцетом, которые позволяют изучать конденсацию одной молекулы ДНК ДНК-связывающим белком. Однако из-за кооперативности жесткие волокна и сети могли формироваться в некоторых областях хромосомы. Само по себе образование филаментов не вызывает конденсации, но сеть или группировка ДНК могут существенно способствовать конденсации, объединяя отдаленные или близлежащие сегменты хромосом.
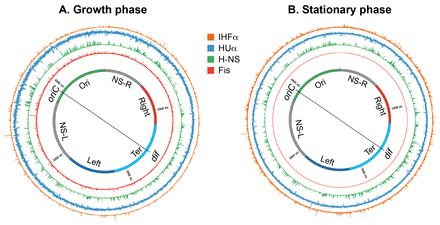 Полногеномная занятость нуклеоид-ассоциированных белков E. coli. Круговая схема генома E. coli, показывающая занятость NAP Fis, H-NS, HU и IHF по всему геному в фазах роста и стационарной фазе у E. coli. Графики гистограммы занятости NAP в геноме, определяемой иммунопреципитацией хроматина в сочетании с секвенированием ДНК (ChIP-seq), показаны вне кольцевого генома. Размер бина гистограмм составляет 300 п.н. Рисунок подготовлен в circos / 0.69-6 с использованием данных ChIP-Seq из.
Полногеномная занятость нуклеоид-ассоциированных белков E. coli. Круговая схема генома E. coli, показывающая занятость NAP Fis, H-NS, HU и IHF по всему геному в фазах роста и стационарной фазе у E. coli. Графики гистограммы занятости NAP в геноме, определяемой иммунопреципитацией хроматина в сочетании с секвенированием ДНК (ChIP-seq), показаны вне кольцевого генома. Размер бина гистограмм составляет 300 п.н. Рисунок подготовлен в circos / 0.69-6 с использованием данных ChIP-Seq из. Фактор хоста интеграции (IHF) структурно почти идентичен HU, но во многих аспектах ведет себя иначе, чем HU. В отличие от HU, который предпочтительно связывается со структурным мотивом независимо от последовательности, IHF предпочтительно связывается со специфической последовательностью ДНК, даже если специфичность возникает из-за зависимой от последовательности структуры ДНК и деформируемости. Специфическое связывание IHF на родственных сайтах резко изгибает ДНК более чем на 160 градусов. Мотив родственной последовательности встречается около 3000 в геноме E. coli. Расчетное содержание IHF в фазе роста составляет около 6000 димеров на клетку. Предполагая, что один димер IHF связывается с одним мотивом, а нуклеоид содержит более одного эквивалента генома во время фазы экспоненциального роста, большинство молекул IHF будет занимать определенные места в геноме и, вероятно, только конденсировать ДНК, вызывая резкий изгиб.
Помимо преимущественного связывания с конкретной последовательностью ДНК, IHF также связывается с ДНК неспецифическим для последовательности образом со сродством, аналогичным HU. Роль неспецифического связывания IHF в конденсации ДНК, по-видимому, является критической в стационарной фазе, поскольку содержание IHF увеличивается в пять раз в стационарной фазе, и дополнительные димеры IHF, вероятно, будут связываться с хромосомной ДНК неспецифично. В отличие от HU, IHF не образует толстых жестких волокон при более высоких концентрациях. Вместо этого его неспецифическое связывание также вызывает изгибание ДНК, хотя степень изгиба намного меньше, чем в определенных сайтах, и подобна гибкому изгибу, индуцированному HU в линейной ДНК при низких концентрациях. In vitro изгиб, вызванный неспецифическим связыванием IHF, может вызывать конденсацию ДНК и способствует образованию комплексов нуклеопротеидов более высокого порядка в зависимости от концентраций хлорида калия и хлорида магния. Организация ДНК более высокого порядка с помощью IHF in vivo пока неясна.
Отличительной особенностью гистоноподобного или термостабильного структурирующего нуклеоид белка (H-NS) от других NAP является способность переключаться с гомодимерной формы при относительно низких концентрациях (lt;1 x 10 -5 M) в олигомерное состояние при более высоких концентрациях. уровни. Из-за свойств олигомеризации H-NS распространяется латерально вдоль AT-богатой ДНК в реакции зародышеобразования, где сайты с высоким сродством функционируют как центры зародышеобразования. Распространение H-NS на ДНК приводит к двум противоположным результатам в зависимости от концентрации магния в реакции. При низкой концентрации магния (lt;2 мМ) H-NS образует жесткие нуклеопротеиновые филаменты, тогда как он образует меж- и внутримолекулярные мостики при более высоких концентрациях магния (gt; 5 мМ). Образование жестких нитей приводит к выпрямлению ДНК без конденсации, тогда как образование мостиков вызывает существенное сворачивание ДНК. Анализ связывания H-NS в геноме с помощью анализов ChIP-Seq предоставил косвенные доказательства распространения H-NS на ДНК in vivo. H-NS избирательно связывается с 458 участками генома. Хотя было продемонстрировано, что H-NS предпочитает изогнутую ДНК, образованную повторяющимися A-треками в последовательностях ДНК, основой избирательного связывания является присутствие консервативного мотива последовательности, обнаруженного в областях, богатых AT. Что еще более важно, частое появление мотива последовательности в области связывания H-NS, которое может усиливать кооперативные взаимодействия белок-белок, и необычно большая длина области связывания согласуются с распространением белка. Преобладает ли образование филаментов или образование мостиков ДНК in vivo, зависит от физиологической концентрации магния внутри клетки. Если концентрация магния равномерно низкая (lt;5 мМ), H-NS будет образовывать жесткие нуклеопротеиновые филаменты in vivo. С другой стороны, если в клетке наблюдается неравномерное распределение магния, это может способствовать как образованию мостиков, так и повышению жесткости ДНК, но в разных областях нуклеоида.
Более того, H-NS наиболее известен как глобальный глушитель, который преимущественно ингибирует транскрипцию горизонтально переносимых генов, и именно жесткий филамент приводит к молчанию генов. Взятые вместе, оказывается, что образование жестких филаментов является наиболее вероятным результатом взаимодействий H-NS-ДНК in vivo, которое приводит к молчанию генов, но не вызывает конденсацию ДНК. Соответственно, отсутствие H-NS не изменяет объем нуклеоида. Однако возможно, что E. coli имеет высокую концентрацию магния в некоторых условиях окружающей среды. В таких условиях H-NS может переключаться из своей индуцирующей филамент формы на индуцирующую мостик форму, которая способствует конденсации и организации ДНК.
Фактор инверсионной стимуляции (Fis) представляет собой специфичный для последовательности ДНК-связывающий белок, который связывается со специфическими последовательностями ДНК, содержащими симметричный мотив длиной 15 пар оснований. Подобно IHF, Fis вызывает изгиб ДНК в родственных участках. Способность изгибать ДНК проявляется в структуре гомодимера Fis. Гомодимер Fis имеет два мотива спираль-поворот-спираль (HTH), по одному от каждого мономера. Мотив HTH обычно распознает большую бороздку ДНК. Однако расстояние между спиралями распознавания ДНК двух мотивов HTH в гомодимере Fis составляет 25 Å, что на ~ 8 Å короче шага канонической B-ДНК, что указывает на то, что белок должен изгибать или скручивать ДНК для стабильного связывания.. Соответственно, кристаллическая структура комплексов Fis-ДНК показывает, что расстояние между спиралями узнавания остается неизменным, тогда как ДНК изгибается в диапазоне 60-75 градусов. В геноме E. coli распределены 1464 связывающих участка Fis, и связывающий мотив, идентифицированный компьютерным путем, совпадает с известным мотивом из 15 пар оснований. Специфическое связывание Fis в таких сайтах будет вызывать изгибы в ДНК, таким образом, способствуя конденсации ДНК за счет уменьшения длины персистентности ДНК. Кроме того, многие сайты связывания Fis встречаются в тандеме, например, в промоторах стабильной РНК, например, промоторе P1 оперона рРНК rrnB. Когерентное изгибание Fis в тандемных сайтах, вероятно, создает микропетлю ДНК, которая может вносить дополнительный вклад в конденсацию ДНК.
Помимо высокоаффинного специфического связывания с родственными сайтами, Fis может связываться со случайной последовательностью ДНК. Неспецифическое связывание ДНК имеет большое значение, поскольку в фазе роста количество Fis равно HU. Следовательно, ожидается, что большинство молекул Fis будут связываться с ДНК неспецифическим для последовательности образом. Эксперименты с магнитным пинцетом показывают, что это неспецифическое связывание Fis может способствовать конденсации и организации ДНК. Fis вызывает мягкую конденсацию одиночной молекулы ДНК при концентрации lt;1 мМ, но вызывает существенное сворачивание за счет образования петель ДНК среднего размера ~ 800 п.н. при концентрацииgt; 1 мМ. Петли в экспериментах с магнитным пинцетом отличаются от микропетлей, созданных когерентным изгибом ДНК на родственных участках, поскольку они требуют образования комплексов ДНК-белок высокой плотности, достигаемых за счет независимого от последовательности связывания. Хотя появление таких петель in vivo еще предстоит продемонстрировать, связывание с высокой плотностью Fis может происходить in vivo за счет согласованного действия как специфического, так и неспецифического связывания. Тандемное возникновение специфических сайтов может инициировать реакцию зародышеобразования, аналогичную реакции H-NS, а затем неспецифическое связывание должно приводить к образованию локализованных массивов Fis с высокой плотностью. Мостик между этими локализованными областями может создавать большие петли ДНК. Fis присутствует исключительно в фазе роста, а не в стационарной фазе. Таким образом, любая роль Fis в хромосомной конденсации должна быть специфичной для растущих клеток.
Ранние исследования, изучающие влияние обработки РНКазой А на изолированные нуклеоиды, показали, что РНК участвует в стабилизации нуклеоида в конденсированном состоянии. Более того, обработка РНКазой А разрушала волокна ДНК на более тонкие волокна, что наблюдалось с помощью атомно-силовой микроскопии нуклеоида с использованием «процедуры лизиса на субстрате». Эти данные продемонстрировали участие РНК в структуре нуклеоида, но идентичность молекулы (молекул) РНК оставалась неизвестной до недавнего времени. Большинство исследований HU сосредоточено на его связывании с ДНК. Однако HU также связывается с гибридами дцРНК и РНК-ДНК с более низким сродством, аналогичным сродству с линейной дцДНК. Более того, HU предпочтительно связывается с РНК, содержащей вторичные структуры, и гибридом РНК-ДНК, в котором РНК содержит разрыв или выступ. Аффинность связывания HU с этими субстратами РНК аналогична аффинности связывания HU с искаженной ДНК. Иммунопреципитация HU-связанной РНК, связанной с обратной транскрипцией и микрочипом (RIP-Chip), а также анализ РНК из очищенных интактных нуклеоидов идентифицировали нуклеоид-ассоциированные молекулы РНК, которые взаимодействуют с HU. Некоторые из них являются некодирующими РНК, и одна такая РНК, названная naRNA4 (нуклеоид-ассоциированная РНК 4), кодируется в повторяющемся экстрагенном палиндроме ( REP325). В штамме без REP325 нуклеоид деконденсируется, как и в штамме без HU. НаРНК4, скорее всего, участвует в конденсации ДНК, соединяя сегменты ДНК в присутствии HU. Недавние исследования дают представление о молекулярном механизме того, как наРНК4 устанавливает связи ДНК-ДНК. РНК нацелена на области ДНК, содержащие крестообразные структуры, и образует комплекс РНК-ДНК, который имеет решающее значение для установления связей ДНК-ДНК. Удивительно, хотя HU помогает в образовании комплекса, он не присутствует в конечном комплексе, что указывает на его потенциальную роль в качестве катализатора (шаперона). Природа комплекса РНК-ДНК остается загадкой, потому что образование комплекса не включает в себя обширное спаривание оснований Уотсона / Крика, но чувствительно к РНКазе H, которая расщепляет РНК в гибриде РНК-ДНК, и комплекс связывается с антителом, специфичным для Гибриды РНК-ДНК.
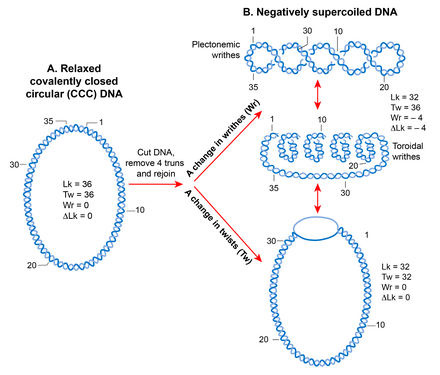 Суперспирализация ДНК A. Линейная двухцепочечная ДНК становится топологически ограниченной молекулой, если два конца ковалентно соединены, образуя круг. Правила топологии ДНК объясняются с использованием такой молекулы (ccc-ДНК), в которой числовой параметр, называемый числом связи (Lk), определяет топологию. Lk представляет собой математическую сумму двух геометрических параметров: скручивания (Tw) и скручивания (Wr). Скручивание - это пересечение двух нитей, а изгиб - это скручивание двойной спирали ДНК вокруг своей оси, которое требует изгиба. Lk всегда является целым числом и остается неизменным независимо от того, насколько деформированы две нити. Его можно изменить только путем введения разрыва в одной или обеих цепях ДНК метаболическими ферментами ДНК, называемыми топоизомеразами. B. Деформация кручения, создаваемая изменением Lk расслабленной, топологически ограниченной ДНК, проявляется в форме суперспирализации ДНК. Уменьшение Lk (Lk lt;Lk 0) вызывает отрицательную суперспирализацию, тогда как увеличение Lk (Lkgt; Lk 0) вызывает положительную суперспирализацию. Здесь изображена только отрицательная суперспирализация. Например, если разрез вводится в ccc-ДНК и четыре витка удаляются перед повторным соединением двух цепей, ДНК приобретает отрицательную суперспирализацию с уменьшением числа скручиваний или корчков, или и того, и другого. Writhe может использовать два типа геометрических структур, называемых плектонемой и тороид. Для плектонем характерно переплетение двойной спирали ДНК и апикальной петли, тогда как спиралевание двойной спирали ДНК вокруг оси образует тороиды.
Суперспирализация ДНК A. Линейная двухцепочечная ДНК становится топологически ограниченной молекулой, если два конца ковалентно соединены, образуя круг. Правила топологии ДНК объясняются с использованием такой молекулы (ccc-ДНК), в которой числовой параметр, называемый числом связи (Lk), определяет топологию. Lk представляет собой математическую сумму двух геометрических параметров: скручивания (Tw) и скручивания (Wr). Скручивание - это пересечение двух нитей, а изгиб - это скручивание двойной спирали ДНК вокруг своей оси, которое требует изгиба. Lk всегда является целым числом и остается неизменным независимо от того, насколько деформированы две нити. Его можно изменить только путем введения разрыва в одной или обеих цепях ДНК метаболическими ферментами ДНК, называемыми топоизомеразами. B. Деформация кручения, создаваемая изменением Lk расслабленной, топологически ограниченной ДНК, проявляется в форме суперспирализации ДНК. Уменьшение Lk (Lk lt;Lk 0) вызывает отрицательную суперспирализацию, тогда как увеличение Lk (Lkgt; Lk 0) вызывает положительную суперспирализацию. Здесь изображена только отрицательная суперспирализация. Например, если разрез вводится в ccc-ДНК и четыре витка удаляются перед повторным соединением двух цепей, ДНК приобретает отрицательную суперспирализацию с уменьшением числа скручиваний или корчков, или и того, и другого. Writhe может использовать два типа геометрических структур, называемых плектонемой и тороид. Для плектонем характерно переплетение двойной спирали ДНК и апикальной петли, тогда как спиралевание двойной спирали ДНК вокруг оси образует тороиды. Из-за своей спиральной структуры молекула двухцепочечной ДНК становится топологически ограниченной в ковалентно замкнутой круговой форме, что исключает вращение свободных концов. Количество раз, когда две нити пересекаются друг с другом в топологически ограниченной ДНК, называется числом связывания (Lk), которое эквивалентно количеству спиральных витков или витков в кольцевой молекуле. Lk топологической ДНК остается неизменной, независимо от того, как деформируется молекула ДНК, до тех пор, пока ни одна из цепей не разорвана.
Lk ДНК в расслабленной форме определяется как Lk 0. Для любой ДНК Lk 0 можно рассчитать путем деления длины (в п.о.) ДНК на количество п.н. на виток спирали. Это равно 10,4 п.н. для релаксированной B-формы ДНК. Любое отклонение от Lk 0 вызывает суперспирализацию ДНК. Уменьшение числа связей (Lk lt;Lk 0) создает отрицательную суперспирали, тогда как увеличение числа связей (Lkgt; Lk 0) создает положительную суперспирацию.
Состояние суперспирали (когда Lk не равно Lk 0) приводит к переходу в структуре ДНК, который может проявляться как изменение числа скручиваний (отрицательное lt;10,4 п.н. / виток, положительноеgt; 10,4 п.н. на оборот) и / или в образование корч, называемых суперспиралями. Таким образом, Lk математически определяется как знакозависимая сумма двух геометрических параметров, скручивания и изгиба. Количественным показателем суперспирализации, не зависящим от размера молекул ДНК, является плотность суперспирализации (σ), где σ = ∆Lk / Lk 0.
Writhes может принимать две структуры; плектонема и соленоид или тороид. Плектонемическая структура возникает из-за переплетения винтовой оси. Тороидальные суперспирали образуются, когда ДНК образует несколько спиралей вокруг оси и не пересекается друг с другом, как в телефонном шнуре. Корки в форме плектонем являются правосторонними и левосторонними в положительно или отрицательно свернутой ДНК соответственно. Направленность тороидальных суперспиралей противоположна таковой у плектонем. И плектонемы, и тороидальные суперспирали могут быть как в свободной форме, так и в связанной форме с помощью белков. Лучшим примером связанной тороидальной суперспирализации в биологии является нуклеосома эукариот, в которой ДНК оборачивается вокруг гистонов.
 Основные единицы геномной организации у бактерий и эукариот. Геномная ДНК, изображенная серой линией, имеет отрицательную суперспираль как у бактерий, так и у эукариот. Однако отрицательно свернутая ДНК организована в плектонемной форме у бактерий, тогда как у эукариот она организована в тороидальной форме. Связанные с нуклеоидом белки (NAP), показанные в виде цветных сфер, сдерживают половину плектонемических суперспиралей, тогда как почти все тороидальные суперспирали индуцируются, а также сдерживаются нуклеосомами (окрашены в оранжевый цвет), образованными путем наматывания ДНК на гистоны.
Основные единицы геномной организации у бактерий и эукариот. Геномная ДНК, изображенная серой линией, имеет отрицательную суперспираль как у бактерий, так и у эукариот. Однако отрицательно свернутая ДНК организована в плектонемной форме у бактерий, тогда как у эукариот она организована в тороидальной форме. Связанные с нуклеоидом белки (NAP), показанные в виде цветных сфер, сдерживают половину плектонемических суперспиралей, тогда как почти все тороидальные суперспирали индуцируются, а также сдерживаются нуклеосомами (окрашены в оранжевый цвет), образованными путем наматывания ДНК на гистоны. У большинства бактерий ДНК присутствует в суперспиральной форме. Круговая природа хромосомы E. coli делает ее топологически ограниченной молекулой, которая в основном имеет отрицательную суперспирали с расчетной средней плотностью суперспирали (σ) -0,05. В эукариотическом хроматине ДНК находится в основном в тороидальной форме, которая ограничивается и определяется гистонами посредством образования нуклеосом. Напротив, в нуклеоиде E. coli около половины хромосомной ДНК организовано в виде свободных плектонемных суперспиралей. Оставшаяся ДНК ограничивается либо плектонемной формой, либо альтернативными формами, включая, но не ограничиваясь, тороидальную форму, посредством взаимодействия с белками, такими как NAP. Таким образом, плектонемические суперспирали представляют собой эффективную суперспирализацию генома E. coli, которая отвечает за его конденсацию и организацию. И плектонемная, и тороидальная суперспирализация способствуют конденсации ДНК. Примечательно, что из-за разветвления плектонемных структур он обеспечивает меньшую конденсацию ДНК, чем тороидальная структура. Молекула ДНК одинакового размера с одинаковой плотностью суперспирали более компактна в тороидальной форме, чем в плектонемной форме. Помимо конденсации ДНК, суперспирализация помогает в организации ДНК. Это способствует распутыванию ДНК за счет снижения вероятности образования цепочки. Суперспирализация также помогает сблизить два удаленных участка ДНК, тем самым способствуя потенциальному функциональному взаимодействию между различными сегментами ДНК.
Три фактора способствуют созданию и поддержанию суперспирализации хромосомной ДНК в E. coli: (i) активность топоизомераз, (ii) акт транскрипции и (iii) NAP.
Топоизомеразы представляют собой особую категорию метаболических ферментов ДНК, которые создают или удаляют суперспирализацию путем разрыва, а затем повторного лигирования цепей ДНК. E. coli содержит четыре топоизомеразы. ДНК-гираза вызывает отрицательную суперспирализацию в присутствии АТФ и удаляет положительную суперспирализацию в отсутствие АТФ. Во всех формах жизни ДНК-гираза является единственной топоизомеразой, которая может создавать отрицательную суперспирализацию, и именно благодаря этой уникальной способности бактериальные геномы обладают свободными отрицательными суперспиралями; ДНК-гираза обнаружена у всех бактерий, но отсутствует у высших эукариот. Напротив, Topo I противостоит ДНК-гиразе, расслабляя отрицательно свернутую ДНК. Имеются генетические данные, позволяющие предположить, что баланс между противоположной активностью ДНК-гиразы и Topo I отвечает за поддержание устойчивого уровня средней отрицательной супервосприимчивости E. coli. Оба фермента необходимы для выживания E. coli. Нулевой штамм topA, гена, кодирующего Topo I, выживает только благодаря наличию супрессорных мутаций в генах, кодирующих ДНК-гиразу. Эти мутации приводят к снижению активности гиразы, предполагая, что избыточная отрицательная суперспирализация из-за отсутствия Topo I компенсируется сниженной активностью отрицательной суперспирализации ДНК-гиразы. Topo III незаменим в E. coli и, как известно, не играет никакой роли в суперспирализации у E. coli. Основная функция Topo IV - разрешить сестринские хромосомы. Однако было показано, что он также вносит свой вклад в установившийся уровень отрицательной суперспирали, расслабляя отрицательную сверхспирали вместе с Topo I.
| Топоизомераза | Тип | Функция | Одно- или двухцепочечное расщепление |
|---|---|---|---|
| Топоизомераза I | Я | Удаляет (-) суперспирализацию | SS |
| Топоизомераза III | Я | Удаляет (-) суперспирализацию | SS |
| Топоизомераза IV | IIA | Удаляет (-) суперспирализацию | DS |
| ДНК-гираза | IIA | Создает (-) суперспирали и удаляет (+) суперспирали | DS |
Модель двойного суперспирального домена, предложенная Лю и Вангом, утверждала, что раскручивание двойной спирали ДНК во время транскрипции вызывает суперспирализацию ДНК, как показано на рисунке. Согласно их модели, транскрибирующая РНК-полимераза (РНКП), скользящая вдоль ДНК, заставляет ДНК вращаться вокруг своей спиральной оси.. Помехи в свободном вращении ДНК могут возникать из-за топологического ограничения, в результате чего ДНК перед RNAP становится чрезмерно скрученной (положительно сверхспиральной), а ДНК позади RNAP становится недостаточно скрученной (отрицательно сверхспиральной). Было обнаружено, что топологическое ограничение не требуется, потому что RNAP генерирует достаточный крутящий момент, который вызывает суперспирализацию даже в линейной матрице ДНК. Если ДНК уже имеет отрицательную суперспираль, это действие расслабляет существующие отрицательные суперспирали, прежде чем вызвать накопление положительных суперспиралей перед RNAP и ввести больше отрицательных суперспиралей позади RNAP. В принципе, ДНК-гираза и Topo I должны удалять избыточные положительные и отрицательные суперспирали соответственно, но если скорость элонгации RNAP превышает оборот двух ферментов, транскрипция способствует установившемуся уровню суперспирализации.
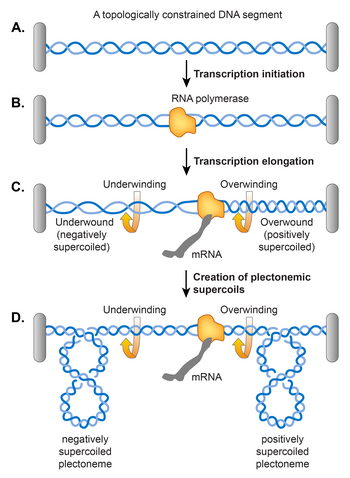 Модель двойного суперспирального домена для индуцированной транскрипцией суперспирализации A. Пример топологически ограниченной ДНК. Серая полоса представляет собой топологическое ограничение, например, белок или якорь мембраны. Б. Аккомодация РНК-полимеразы для инициации транскрипции приводит к раскрытию двойной спирали ДНК. C. Удлиняющийся комплекс РНК-полимеразы не может вращаться вокруг спиральной оси ДНК. Следовательно, удаление витков спирали с помощью РНК-полимеразы вызывает перемотку топологически ограниченной ДНК впереди и перемотку ДНК сзади, генерируя положительно и отрицательно суперспиральную ДНК, соответственно. Суперспирализация может проявляться либо в изменении числа скручиваний, как показано на C, либо как плектонемическая корка, как показано на D.
Модель двойного суперспирального домена для индуцированной транскрипцией суперспирализации A. Пример топологически ограниченной ДНК. Серая полоса представляет собой топологическое ограничение, например, белок или якорь мембраны. Б. Аккомодация РНК-полимеразы для инициации транскрипции приводит к раскрытию двойной спирали ДНК. C. Удлиняющийся комплекс РНК-полимеразы не может вращаться вокруг спиральной оси ДНК. Следовательно, удаление витков спирали с помощью РНК-полимеразы вызывает перемотку топологически ограниченной ДНК впереди и перемотку ДНК сзади, генерируя положительно и отрицательно суперспиральную ДНК, соответственно. Суперспирализация может проявляться либо в изменении числа скручиваний, как показано на C, либо как плектонемическая корка, как показано на D. В хроматине эукариот ДНК редко присутствует в свободной суперспиральной форме, потому что нуклеосомы сдерживают почти все отрицательные суперспирали за счет прочного связывания ДНК с гистонами. Точно так же в E. coli нуклеопротеиновые комплексы, образованные NAP, сдерживают половину плотности суперспирализации нуклеоида. Другими словами, если NAP отделяется от нуклеопротеинового комплекса, ДНК принимает свободную плектонемную форму. Связывание ДНК HU, Fis и H-NS было экспериментально показано, чтобы сдерживать отрицательную суперспирализацию в расслабленной, но топологически ограниченной ДНК. Они могут делать это либо изменяя шаг спирали ДНК, либо создавая тороидальные корки за счет изгиба и сворачивания ДНК. Альтернативно, NAP могут предпочтительно связываться и стабилизировать другие формы поврежденной ДНК, такие как крестообразные структуры и разветвленные плектонемы. Сообщалось, что Fis организует разветвленные плектонемы посредством связывания с перекрестными областями, а HU предпочтительно связывается с крестообразными структурами.
NAP также косвенно регулируют сверхспирализацию ДНК. Fis может модулировать суперспирализацию путем подавления транскрипции генов, кодирующих ДНК-гиразу. Имеются генетические данные, позволяющие предположить, что HU контролирует уровни суперспирализации путем стимуляции ДНК-гиразы и снижения активности Topo I. В поддержку генетических исследований было показано, что HU стимулирует декатенацию ДНК, катализируемую ДНК-гиразой, in vitro. Механически неясно, как HU модулирует активность гиразы и Topo I. HU может физически взаимодействовать с ДНК-гиразой и Topo I, или активность HU по организации ДНК, такая как изгиб ДНК, может облегчать или ингибировать действие ДНК-гиразы и Topo I соответственно.
Одна из поразительных особенностей нуклеоида состоит в том, что плектонемические суперспирали организованы в несколько топологических доменов. Другими словами, одно сокращение в одной области ослабит только эту область, а не другие. Топологическая область образуется из-за барьера суперспирализации-диффузии. Независимые исследования с использованием различных методов показали, что размер топологических доменов варьируется от 10 до 400 т.п.н. Случайное размещение барьеров, обычно наблюдаемое в этих исследованиях, по-видимому, объясняет широкий разброс размеров доменов.
Хотя идентичность доменных барьеров еще предстоит установить, возможные механизмы, ответственные за формирование барьеров, включают: (i) Доменный барьер может формироваться, когда белок, способный сдерживать суперспирали, одновременно связывается с двумя разными сайтами на хромосоме, формируя топологически изолированная петля или домен ДНК. Экспериментально продемонстрировано, что опосредованное белком образование петель в суперспиральной ДНК может создавать топологический домен. NAP, такие как H-NS и Fis, являются потенциальными кандидатами, исходя из их способности образовывать петли ДНК и распределения их сайтов связывания. (ii) Бактериальные вкрапленные мозаичные элементы (BIME) также являются потенциальными кандидатами на роль доменных барьеров. BIME представляют собой последовательности палиндромных повторов, которые обычно встречаются между генами. Было показано, что BIME препятствует распространению суперспирали в синтетически сконструированной топологической кассете, вставленной в хромосому E. coli. По геному распределено ~ 600 BIME, которые, возможно, делят хромосому на 600 топологических доменов. (iii) Барьеры также могут возникать в результате прикрепления ДНК к клеточной мембране через белок, который связывается как с ДНК, так и с мембраной, или за счет зарождающейся транскрипции и трансляции закрепленных на мембране белков. (iv) Транскрипционная активность может создавать барьеры суперспирализации-диффузии. Было показано, что активно транскрибирующий RNAP блокирует диссипацию плектонемных суперспиралей, тем самым формируя барьер суперспирализации-диффузии.
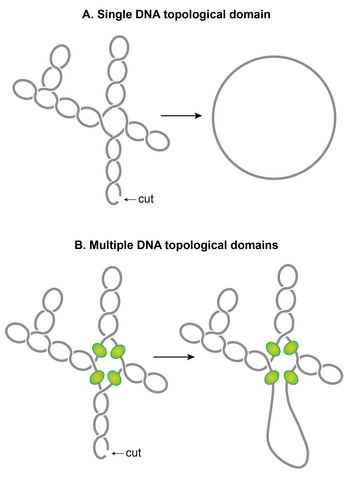 Хромосомная ДНК внутри нуклеоида разделена на независимые суперспиральные топологические домены A. Иллюстрация единственного топологического домена суперспиральной ДНК. Одного двухниточного разреза в любом месте было бы достаточно, чтобы ослабить натяжение суперспирали всего домена. Б. Иллюстрация нескольких топологических доменов в сверхспиральной молекуле ДНК. Наличие барьеров суперспирализации-диффузии разделяет сверхспиральную молекулу ДНК на несколько топологических доменов. Гипотетические диффузионные барьеры сверхспирализации представлены зелеными сферами. В результате одинарный двухцепочечный разрез ослабит только одну топологическую область, а не остальные. Плектонемические суперспирали ДНК внутри нуклеоида E. coli организованы в несколько топологических доменов, но для простоты показаны только четыре домена с разным количеством суперспиралей.
Хромосомная ДНК внутри нуклеоида разделена на независимые суперспиральные топологические домены A. Иллюстрация единственного топологического домена суперспиральной ДНК. Одного двухниточного разреза в любом месте было бы достаточно, чтобы ослабить натяжение суперспирали всего домена. Б. Иллюстрация нескольких топологических доменов в сверхспиральной молекуле ДНК. Наличие барьеров суперспирализации-диффузии разделяет сверхспиральную молекулу ДНК на несколько топологических доменов. Гипотетические диффузионные барьеры сверхспирализации представлены зелеными сферами. В результате одинарный двухцепочечный разрез ослабит только одну топологическую область, а не остальные. Плектонемические суперспирали ДНК внутри нуклеоида E. coli организованы в несколько топологических доменов, но для простоты показаны только четыре домена с разным количеством суперспиралей. Нуклеоид реорганизуется в клетках со стационарной фазой, предполагая, что структура нуклеоида очень динамична и определяется физиологическим состоянием клеток. Сравнение карт контактов нуклеоида с высоким разрешением показало, что дальнодействующие контакты в макродомене Ter увеличиваются в стационарной фазе по сравнению с фазой роста. Кроме того, границы CID в стационарной фазе отличались от границ, обнаруженных в фазе роста. Наконец, морфология нуклеоидов претерпевает массивную трансформацию во время длительной стационарной фазы; нуклеоид имеет упорядоченную тороидальную структуру.
Специфические для фазы роста изменения в структуре нуклеоидов могут быть вызваны изменением уровней связанных с нуклеоидами архитектурных белков ДНК (NAPs и субъединицы Muk), суперспирализации и транскрипционной активности. Количество NAP и субъединиц Muk изменяется в соответствии с циклом роста бактерий. Fis и ДНК-связывающий белок Dps, индуцированный голоданием, другой NAP, почти исключительно присутствуют в фазе роста и стационарной фазе соответственно. Уровни Fis повышаются при входе в экспоненциальную фазу, а затем быстро снижаются, пока клетки все еще находятся в экспоненциальной фазе, достигая уровней, которые невозможно обнаружить в стационарной фазе. В то время как уровни Fis начинают снижаться, уровни Dps начинают расти и достигают максимума в стационарной фазе. Резкий переход в структуре нуклеоида, наблюдаемый в длительной стационарной фазе, в основном приписывается Dps. Он образует ДНК / кристаллические сборки, которые защищают нуклеоид от агентов, повреждающих ДНК, присутствующих во время голодания.
HU, IHF и H-NS присутствуют как в фазе роста, так и в стационарной фазе. Однако их численность значительно изменяется, так что HU и Fis являются наиболее распространенными NAP в фазе роста, тогда как IHF и Dps становятся наиболее распространенными NAP в стационарной фазе. HUαα является преобладающей формой в ранней экспоненциальной фазе, тогда как гетеродимерная форма преобладает в стационарной фазе с небольшими количествами гомодимеров. Этот переход имеет функциональные последствия в отношении структуры нуклеоидов, потому что эти две формы, по-видимому, по-разному организуют и конденсируют ДНК; как гомо-, так и гетеродимеры образуют филаменты, но только гомодимер может объединять несколько сегментов ДНК с образованием сети ДНК. Число копий MukB увеличивается вдвое в стационарной фазе. Увеличение количества молекул MukB может влиять на процессивность комплекса MukBEF как фактора экструдирования петли ДНК, приводя к большему или большему количеству петель.
Суперспирализация может действовать согласованно с архитектурными белками ДНК для реорганизации нуклеоида. Общий уровень суперспирализации уменьшается в стационарной фазе, и суперспирализация демонстрирует иную картину на региональном уровне. Изменения суперспирализации могут изменить топологическую организацию нуклеоида. Более того, поскольку хромосомная область с высокой транскрипционной активностью формирует границу CID, изменения транскрипционной активности во время разных фаз роста могут изменять формирование границ CID и, таким образом, пространственную организацию нуклеоида. Возможно, что изменения границ CID, наблюдаемые в стационарной фазе, могут быть связаны с высокой экспрессией другого набора генов в стационарной фазе по сравнению с фазой роста.
Структура хромосомы E. coli и экспрессия генов, по-видимому, взаимно влияют друг на друга. С одной стороны, корреляция границы CID с высокой транскрипционной активностью указывает на то, что организация хромосом управляется транскрипцией. С другой стороны, трехмерная структура ДНК внутри нуклеоида на любом уровне может быть связана с экспрессией генов. Во-первых, было показано, что реорганизация трехмерной архитектуры нуклеоида в E. coli может динамически модулировать паттерн клеточной транскрипции. Мутант HUa сделал нуклеоид очень сильно конденсированным за счет повышенной положительной суперпрочности хромосомной ДНК. Следовательно, многие гены были репрессированы, и многие «покоящиеся» гены были экспрессированы. Кроме того, существует множество конкретных случаев, когда локальные архитектурные изменения, опосредованные белком, изменяют транскрипцию генов. Например, образование жестких нуклеопротеиновых филаментов с помощью H-NS блокирует доступ RNAP к промотору, таким образом предотвращая транскрипцию гена. Посредством сайленсинга генов H-NS действует как глобальный репрессор, предпочтительно ингибируя транскрипцию горизонтально переносимых генов. В другом примере специфическое связывание HU с гал- опероном способствует образованию петли ДНК, которая поддерживает репрессию гал- оперона в отсутствие индуктора. Топологически отличная микропетля ДНК, созданная когерентным изгибом ДНК под действием Fis на стабильных промоторах РНК, активирует транскрипцию. Изгибание ДНК под действием IHF по-разному контролирует транскрипцию с двух тандемных промоторов оперона ilvGMEDA в E. coli. Специфические топологические изменения NAP не только регулируют транскрипцию генов, но также участвуют в других процессах, таких как инициация репликации ДНК, рекомбинация и транспозиция. В отличие от специфической генной регуляции, еще предстоит выяснить, как структура хромосом более высокого порядка и ее динамика влияют на экспрессию генов в глобальном масштабе на молекулярном уровне.
Между суперспирализацией ДНК и транскрипцией генов существует двусторонняя взаимосвязь. Отрицательная суперспирализация промоторной области может стимулировать транскрипцию, облегчая плавление промотора и увеличивая аффинность связывания ДНК белкового регулятора. Стохастические всплески транскрипции, по-видимому, являются общей характеристикой высоко экспрессируемых генов, а уровни суперспирализации матрицы ДНК вносят вклад в взрыв транскрипции. Согласно модели двойных суперспиральных доменов, транскрипция гена может влиять на транскрипцию других близлежащих генов через реле суперспирализации. Одним из таких примеров является активация промотора leu-500. Суперспирализация не только опосредует специфические для генов изменения, но также опосредует крупномасштабные изменения в экспрессии генов. Топологическая организация нуклеоида может позволить независимую экспрессию чувствительных к суперспирализации генов в различных топологических доменах. Карта неограниченной суперспирализации в масштабе генома показала, что области генома имеют разные стационарные плотности суперспирализации, что указывает на то, что уровень суперспирализации отличается в отдельных топологических доменах. В результате изменение суперспирали может привести к доменной экспрессии генов, в зависимости от уровня суперспирализации в каждом домене.
Эффект суперспирализации на экспрессию генов может быть опосредован NAP, которые прямо или косвенно влияют на суперспирализацию. Влияние HU на экспрессию генов, по-видимому, включает изменение суперспирализации и, возможно, организацию ДНК более высокого порядка. Положительная корреляция между связыванием ДНК-гиразы и активацией генов, вызванной отсутствием HU, предполагает, что изменения в суперспирализации ответственны за дифференциальную экспрессию. HU, как было обнаружено, также ответственен за позиционный эффект на экспрессию генов за счет изоляции единиц транскрипции путем ограничения индуцированной транскрипцией суперспирализации. Точечные мутации в HUa резко изменили профиль экспрессии генов E. coli, изменив его морфологию, физиологию и метаболизм. В результате мутантный штамм оказался более инвазивным по отношению к клеткам млекопитающих. Этот драматический эффект сопровождался уплотнением нуклеоидов и усилением положительной суперспирализации. Мутантный белок представлял собой октамер, в отличие от димера дикого типа. Он оборачивает ДНК на своей поверхности правосторонним образом, ограничивая положительные суперспирали в отличие от HU дикого типа. Эти исследования показывают, что аминокислотные замены в HU могут оказывать сильное влияние на структуру нуклеоидов, что, в свою очередь, приводит к значительным фенотипическим изменениям.
Поскольку MukB и HU стали критически важными участниками дальнодействующих взаимодействий ДНК, стоит сравнить влияние каждого из этих двух белков на глобальную экспрессию генов. Хотя HU, по-видимому, контролирует экспрессию генов, модулируя плотность суперспирализации, точный молекулярный механизм остается неизвестным, и влияние MukB на экспрессию генов еще предстоит проанализировать.
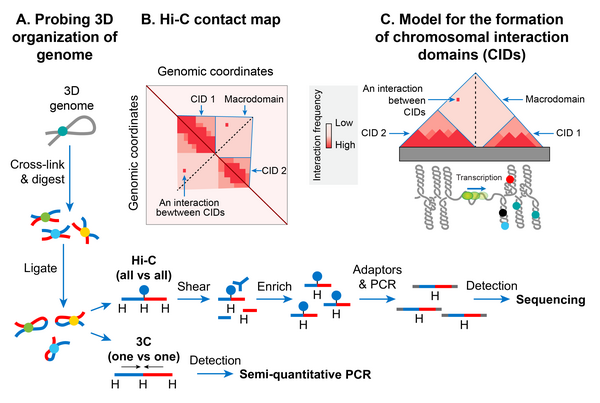 Нуклеоид пространственно организован в домены хромосомных взаимодействий (CID) и макродомены A. Методы захвата конформации хромосомы (3C) исследуют трехмерную организацию генома путем количественной оценки физических взаимодействий между геномными локусами, которые находятся поблизости в трехмерном пространстве, но могут быть далеко в линейном геноме. Геном перекрестно сшивается формальдегидом для сохранения физических контактов между геномными локусами. Впоследствии геном переваривается рестрикционным ферментом. На следующем этапе лигирование ДНК выполняется при разбавленных концентрациях ДНК, чтобы способствовать внутримолекулярному лигированию (между сшитыми фрагментами, которые физически сближаются с трехмерной организацией генома). Частота лигирования между удаленными участками ДНК отражает физическое взаимодействие. В методе 3C соединения лигирования обнаруживаются с помощью полуколичественной ПЦР-амплификации, в которой эффективность амплификации является грубой оценкой попарного физического контакта между интересующими областями генома и его частоты. Метод 3C исследует физическое взаимодействие между двумя определенными областями, идентифицированными априори, тогда как его версия Hi-C обнаруживает физические взаимодействия между всеми возможными парами областей генома одновременно. В методе Hi-C расщепленные концы заполняют биотинилированным адаптером перед лигированием. Лигированные фрагменты разрезаются и затем обогащаются биотином. Затем соединения лигирования обнаруживаются и количественно оцениваются методами секвенирования парных концов следующего поколения. B. Данные Hi-C обычно представлены в виде двумерной матрицы, в которой оси x и y представляют координаты генома. Геном обычно делится на блоки фиксированного размера, например 5 т.п.н. Размер ячеек существенно определяет разрешение контакта. Каждая запись в матрице, m ij, представляет количество считываний химерного секвенирования, сопоставленных с геномными локусами в ячейках i и j. Количественная оценка считываний (представленная в виде тепловой карты) обозначает относительную частоту контактов между геномными локусами ячеек i и j. Отличительной особенностью тепловой карты является диагональная линия, которая появляется из-за более частого физического взаимодействия между локусами, которые очень близки друг к другу в линейном геноме. Интенсивность дальше от диагональной линии представляет относительную частоту физического взаимодействия между локусами, которые находятся далеко друг от друга в линейном геноме. Треугольники высокой интенсивности вдоль диагональной линии представляют сильно самовзаимодействующие домены хромосомных взаимодействий (CID), которые разделены граничной областью, состоящей из меньшего количества взаимодействий. C. У многих видов бактерий, включая E. coli, суперспиральные топологические домены организуются как CID. Плектонемическая суперспирализация способствует высокому уровню взаимодействия между геномными локусами внутри CID, а область без плектонем (PFR), созданная из-за высокой транскрипционной активности, действует как граница CID. Связанные с нуклеоидом белки, изображенные закрашенными кружками, стабилизируют взаимодействия, опосредованные суперспирализацией. Активно транскрибирующая РНК-полимераза (обозначенная зеленой сферой) в PFR блокирует рассеяние суперспирализации между двумя доменами, таким образом, действует как диффузионный барьер суперспирализации. Размер CID колеблется от 30 до 400 кб. Несколько треугольников (CID) сливаются, образуя больший треугольник, представляющий макродомен. Другими словами, CID макродомена физически взаимодействуют друг с другом чаще, чем с CID соседнего макродомена или с геномными локусами вне этого макродомена. Макродомен может содержать несколько CID. Для простоты показан макродомен, содержащий только два CID.
Нуклеоид пространственно организован в домены хромосомных взаимодействий (CID) и макродомены A. Методы захвата конформации хромосомы (3C) исследуют трехмерную организацию генома путем количественной оценки физических взаимодействий между геномными локусами, которые находятся поблизости в трехмерном пространстве, но могут быть далеко в линейном геноме. Геном перекрестно сшивается формальдегидом для сохранения физических контактов между геномными локусами. Впоследствии геном переваривается рестрикционным ферментом. На следующем этапе лигирование ДНК выполняется при разбавленных концентрациях ДНК, чтобы способствовать внутримолекулярному лигированию (между сшитыми фрагментами, которые физически сближаются с трехмерной организацией генома). Частота лигирования между удаленными участками ДНК отражает физическое взаимодействие. В методе 3C соединения лигирования обнаруживаются с помощью полуколичественной ПЦР-амплификации, в которой эффективность амплификации является грубой оценкой попарного физического контакта между интересующими областями генома и его частоты. Метод 3C исследует физическое взаимодействие между двумя определенными областями, идентифицированными априори, тогда как его версия Hi-C обнаруживает физические взаимодействия между всеми возможными парами областей генома одновременно. В методе Hi-C расщепленные концы заполняют биотинилированным адаптером перед лигированием. Лигированные фрагменты разрезаются и затем обогащаются биотином. Затем соединения лигирования обнаруживаются и количественно оцениваются методами секвенирования парных концов следующего поколения. B. Данные Hi-C обычно представлены в виде двумерной матрицы, в которой оси x и y представляют координаты генома. Геном обычно делится на блоки фиксированного размера, например 5 т.п.н. Размер ячеек существенно определяет разрешение контакта. Каждая запись в матрице, m ij, представляет количество считываний химерного секвенирования, сопоставленных с геномными локусами в ячейках i и j. Количественная оценка считываний (представленная в виде тепловой карты) обозначает относительную частоту контактов между геномными локусами ячеек i и j. Отличительной особенностью тепловой карты является диагональная линия, которая появляется из-за более частого физического взаимодействия между локусами, которые очень близки друг к другу в линейном геноме. Интенсивность дальше от диагональной линии представляет относительную частоту физического взаимодействия между локусами, которые находятся далеко друг от друга в линейном геноме. Треугольники высокой интенсивности вдоль диагональной линии представляют сильно самовзаимодействующие домены хромосомных взаимодействий (CID), которые разделены граничной областью, состоящей из меньшего количества взаимодействий. C. У многих видов бактерий, включая E. coli, суперспиральные топологические домены организуются как CID. Плектонемическая суперспирализация способствует высокому уровню взаимодействия между геномными локусами внутри CID, а область без плектонем (PFR), созданная из-за высокой транскрипционной активности, действует как граница CID. Связанные с нуклеоидом белки, изображенные закрашенными кружками, стабилизируют взаимодействия, опосредованные суперспирализацией. Активно транскрибирующая РНК-полимераза (обозначенная зеленой сферой) в PFR блокирует рассеяние суперспирализации между двумя доменами, таким образом, действует как диффузионный барьер суперспирализации. Размер CID колеблется от 30 до 400 кб. Несколько треугольников (CID) сливаются, образуя больший треугольник, представляющий макродомен. Другими словами, CID макродомена физически взаимодействуют друг с другом чаще, чем с CID соседнего макродомена или с геномными локусами вне этого макродомена. Макродомен может содержать несколько CID. Для простоты показан макродомен, содержащий только два CID. В последние годы появление молекулярного метода, называемого захватом конформации хромосом (3C), позволило изучить пространственную организацию хромосом с высоким разрешением как у бактерий, так и у эукариот. 3C и его версия в сочетании с глубоким секвенированием (Hi-C) определяют физическую близость, если таковая имеется, между любыми двумя геномными локусами в трехмерном пространстве. Карта контактов бактериальных хромосом с высоким разрешением, включая хромосому E. coli, показала, что бактериальная хромосома сегментирована на множество сильно самовзаимодействующих областей, называемых доменами взаимодействия хромосом (CID). CID эквивалентны топологически ассоциированным доменам (TAD), наблюдаемым во многих эукариотических хромосомах, что позволяет предположить, что образование CID является общим феноменом организации генома. Две характеристики определяют CID или TAD. Во-первых, геномные области CID физически взаимодействуют друг с другом чаще, чем с геномными областями за пределами этого CID или с регионами соседнего CID. Во-вторых, наличие границы между CID, которая предотвращает физические взаимодействия между областями генома двух соседних CID.
Было обнаружено, что хромосома E. coli состоит из 31 CID в фазе роста. Размер CID варьировался от 40 до ~ 300 кб. Похоже, что барьер суперспирализации-диффузии, ответственный за разделение плектонемных петель ДНК на топологические домены, функционирует как граница CID у E. coli и многих других бактерий. Другими словами, наличие барьера суперспирализации-диффузии определяет образование ХКИ. Результаты исследования хромосом с помощью Hi-C в E. coli, Caulobacter crescentus и Bacillus subtilis сходятся на модели, в которой формируются CID, потому что плектонемическая петля вместе с активностью NAP по организации ДНК способствует физическим взаимодействиям между геномными локусами, а граница CID состоит из область, свободная от плектонем (PFR), которая предотвращает эти взаимодействия. PFR создается из-за высокой транскрипционной активности, потому что спиральное раскручивание ДНК путем активной транскрипции RNAP сдерживает плектонемные суперспирали. В результате также блокируется диссипация суперспиралей, создавая барьер суперспирализации-диффузии. Косвенное доказательство этой модели происходит из наблюдения, что CID бактериальных хромосом, включая хромосому E. coli, демонстрируют высокотранскрибируемые гены на своих границах, что указывает на роль транскрипции в формировании границы CID. Более прямые доказательства были получены из открытия, что размещение гена с высокой степенью транскрибирования в месте, где не было границ, создало новую границу CID в хромосоме C. crescentus. Однако не все границы CID коррелируют с высокотранскрибируемыми генами в хромосоме E. coli, что позволяет предположить, что другие неизвестные факторы также ответственны за формирование границ CID и диффузионных барьеров суперспирализации.
Плектонемические петли ДНК, организованные как топологические домены или CID, по-видимому, сливаются в дальнейшем с образованием больших пространственно различных доменов, называемых макродоменами (MD). В E. coli MD были первоначально идентифицированы как большие сегменты генома, чьи ДНК-маркеры локализованы вместе (совместно локализованы) в исследованиях флуоресцентной гибридизации in situ (FISH). Большая геномная область (~ 1-Mb), покрывающая локус oriC ( ориджин репликации хромосомы), локализована совместно и была названа макродоменом Ori. Аналогичным образом, большая геномная область (~ 1-Mb), покрывающая конечную область репликации ( ter), локализована совместно и была названа макродоменом Ter. Позднее были идентифицированы MD на основании того, как часто пары атт- сайтов лямбда, которые были вставлены в различные отдаленные участки хромосомы, рекомбинировали друг с другом. В этом методе, основанном на рекомбинации, MD был определен как большая область генома, сайты ДНК которой могут в первую очередь рекомбинировать друг с другом, но не с сайтами за пределами этого MD. Метод, основанный на рекомбинации, подтвердил MD Ori и Ter, которые были идентифицированы в исследованиях FISH, и идентифицировал два дополнительных MD.
Два дополнительных MD были образованы дополнительными областями размером ~ 1 Mb, фланкирующими Ter, и были названы Left и Right. Эти четыре MD (Ori, Ter, Left и Right) составляли большую часть генома, за исключением двух областей генома, фланкирующих Ori. Эти две области (NS-L и NS-R) были более гибкими и неструктурированными по сравнению с MD, поскольку сайты ДНК в них рекомбинировали с сайтами ДНК, расположенными в MD с обеих сторон. Генетическое положение oriC, по- видимому, диктует формирование MD, поскольку репозиционирование oriC посредством генетических манипуляций приводит к реорганизации MD. Например, ближайшие к oriC участки генома всегда ведут себя как NS независимо от последовательности ДНК, а более отдаленные участки всегда ведут себя как MD.
Техника Hi-C дополнительно подтвердила иерархическую пространственную организацию CID в форме макродоменов. Другими словами, CID макродомена физически взаимодействовали друг с другом чаще, чем с CID соседнего макродомена или с геномными локусами вне этого макродомена. Данные Hi-C показали, что хромосома E. coli делится на два отдельных домена. Область, окружающая ter, образовывала изолированный домен, который перекрывался с ранее идентифицированным Ter MD. Контакты ДНК-ДНК в этом домене имели место только в диапазоне ~ 280 т.п.н. Остальная часть хромосомы образовывала единый домен, в геномных локусах которого наблюдались контакты в диапазонеgt; 280 т.п.н. В то время как большинство контактов в этом домене были ограничены максимальным расстоянием ~ 500 т.п.н., были две рыхлые области, геномные локусы которых образовывали контакты на еще больших расстояниях (до ~ 1 МП). Эти рыхлые области соответствовали ранее идентифицированным гибким и менее структурированным областям (NS). Границы изолированного домена, охватывающего ter, и две свободные области, идентифицированные методом Hi-C, сегментировали всю хромосому на шесть областей, которые соответствуют четырем MD и двум областям NS, определенным с помощью тестов на основе рекомбинации.
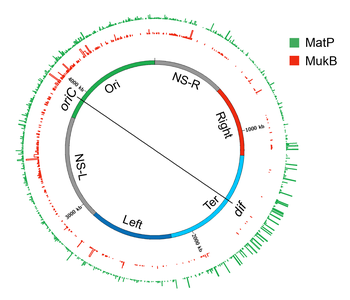 Заселенность MatP и MukB E. coli по всему геному. Круговая структура генома E. coli, показывающая заселенность MatP и MukB в E. coli по всему геному. В самом внутреннем кружке изображен геном E. coli. Области генома, которые организуются как пространственные домены (макродомены) в нуклеоиде, обозначены цветными полосами. Графики гистограммы занятости генома для MatP и MukB, определенной с помощью иммунопреципитации хроматина в сочетании с секвенированием ДНК (ChIP-seq), показаны в кружках. Размер бина гистограмм составляет 300 п.н. Рисунок был подготовлен в circos / 0.69-6 с использованием обработанных данных ChIP-Seq из.
Заселенность MatP и MukB E. coli по всему геному. Круговая структура генома E. coli, показывающая заселенность MatP и MukB в E. coli по всему геному. В самом внутреннем кружке изображен геном E. coli. Области генома, которые организуются как пространственные домены (макродомены) в нуклеоиде, обозначены цветными полосами. Графики гистограммы занятости генома для MatP и MukB, определенной с помощью иммунопреципитации хроматина в сочетании с секвенированием ДНК (ChIP-seq), показаны в кружках. Размер бина гистограмм составляет 300 п.н. Рисунок был подготовлен в circos / 0.69-6 с использованием обработанных данных ChIP-Seq из. Поиск белков, ответственных за образование макродоменов, привел к идентификации белка Macrodomain Ter (MatP). MatP почти исключительно связывается с Ter MD, узнавая мотив длиной 13 пар оснований, называемый последовательностью макродомена ter ( matS). В домене Ter присутствует 23 сайта matS, в среднем по одному сайту каждые 35 кб. Дальнейшие доказательства связывания MatP в домене Ter получены с помощью флуоресцентной визуализации MatP. Наблюдались отдельные фокусы MatP, которые локализовались совместно с ДНК-маркерами Ter-домена. Сильное обогащение сигнала ChIP-Seq в Ter MD также подтверждает предпочтительное связывание MatP с этим доменом.
MatP конденсирует ДНК в домене Ter, поскольку отсутствие MatP увеличивает расстояние между двумя флуоресцентными ДНК-маркерами, расположенными на расстоянии 100 т.п.н. в домене Ter. Кроме того, MatP играет важную роль в изоляции Ter-домена от остальной хромосомы. Он способствует контактам ДНК-ДНК внутри Ter-домена, но предотвращает контакты между ДНК-локусами Ter-домена и локусами фланкирующих областей. Как MatP конденсирует ДНК и способствует контактам ДНК-ДНК? Результаты экспериментов противоречивы. MatP может образовывать петлю ДНК между двумя сайтами matS in vitro, и его активность по образованию петель ДНК зависит от тетрамеризации MatP. Тетрамеризация происходит посредством взаимодействия спиральной спирали между двумя молекулами MatP, связанными с ДНК. Одна очевидная модель, основанная на результатах in vitro, заключается в том, что MatP способствует контактам ДНК-ДНК in vivo посредством связывания сайтов matS. Однако, хотя MatP подключал удаленные сайты в исследованиях Hi-C, он не связывал специально сайты matS. Более того, мутант MatP, который был неспособен образовывать тетрамеры, вел себя как дикий тип. Эти результаты являются аргументом против matS- мостиковой модели для организации Ter, оставляя механизм действия MatP неуловимым. Одна возможность состоит в том, что MatP распространяется на близлежащие сегменты ДНК из своего первичного сайта связывания matS и соединяет отдаленные сайты посредством механизма, который не зависит от тетрамеризации.
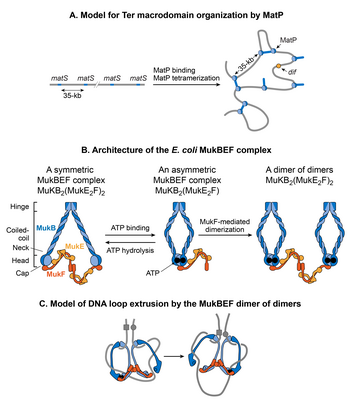 Модели для организации ДНК по MATP и MukBEF A. A Матс -bridging модели организации ДНК в макродомена Ter по MATP. MatP распознает сигнатурную ДНК-последовательность из 13 пар оснований, называемую matS, которая присутствует исключительно в макродомене Ter. Имеется 23 сайта matS, разделенных друг от друга в среднем на 35 т.п.н. MatP связывается с сайтом matS как димер, и тетрамеризация связанных с ДНК димеров связывает сайты matS, образуя большие петли ДНК. Б. Архитектура комплекса E. coli MukBEF. Комплекс образуется в результате белок-белковых взаимодействий между MukB (синий), MukF (темно-оранжевый) и MukE (светло-оранжевый). MukB, который принадлежит к семейству белков структурной поддержки хромосом (SMC), образует димер (мономеры показаны темным и голубым цветами), состоящий из головного домена АТФазы и внутримолекулярной спиральной спирали длиной 100 нм с шарнирной областью. в центре. Благодаря гибкости шарнирной области MukB принимает характерную V-образную форму семейства SMC. MukF также имеет тенденцию существовать в виде димера из-за сильного сродства к димеризации между мономерами. С-концевой домен MukF может взаимодействовать с головным доменом MukB, тогда как его центральный домен может взаимодействовать с MukE. Две молекулы MukE и одна молекула MukF связываются друг с другом независимо от MukB с образованием тримерного комплекса (MukE 2 F). Поскольку MukF имеет тенденцию существовать в димерной форме, димеризация MukF приводит к удлиненному гексамерному комплексу (MukE 2 F) 2. В отсутствие АТФ комплекс (MukE 2 F) 2 связывается с головными доменами MukB через С-концевой домен MukF с образованием симметричного комплекса MukBEF (показан слева). Стехиометрия симметричного комплекса B 2 (E 2 F) 2. Связывание АТФ между головными доменами MukB вызывает отщепление одной молекулы MukF и двух молекул MukE. В результате образуется асимметричный комплекс MukBEF стехиометрии B 2 (E 2 F) 1. Поскольку MukF легко димеризуется, димеризация MukF может потенциально соединять две асимметричные молекулы, связанные с АТФ, что приводит к образованию димера димеров со стехиометрией B 4 (E 2 F) 2 (показано справа). Стехиометрия комплекса MukBEF in vivo оценивается как B 4 (E 2 F) 2, что позволяет предположить, что димер димеров является функциональной единицей in vivo. C. Модель экструзии петель димером димеров MukBEF. Димер димера загружается на ДНК (изображена серой линией) через ДНК-связывающие домены MukB. Было показано, что MukB связывает ДНК через свою шарнирную область и верхнюю область своего головного домена. Транслокация комплекса от места его загрузки затем вытесняет петли ДНК. Петли выдавливаются по-скалолазным образом за счет скоординированного открытия и закрытия кольца MukBEF через разъединение головки MukB, которое происходит из-за скоординированного гидролиза АТФ в двух димерах. Темные и голубые круги обозначают события связывания и гидролиза АТФ соответственно. MukE не показан в комплексе для простоты.
Модели для организации ДНК по MATP и MukBEF A. A Матс -bridging модели организации ДНК в макродомена Ter по MATP. MatP распознает сигнатурную ДНК-последовательность из 13 пар оснований, называемую matS, которая присутствует исключительно в макродомене Ter. Имеется 23 сайта matS, разделенных друг от друга в среднем на 35 т.п.н. MatP связывается с сайтом matS как димер, и тетрамеризация связанных с ДНК димеров связывает сайты matS, образуя большие петли ДНК. Б. Архитектура комплекса E. coli MukBEF. Комплекс образуется в результате белок-белковых взаимодействий между MukB (синий), MukF (темно-оранжевый) и MukE (светло-оранжевый). MukB, который принадлежит к семейству белков структурной поддержки хромосом (SMC), образует димер (мономеры показаны темным и голубым цветами), состоящий из головного домена АТФазы и внутримолекулярной спиральной спирали длиной 100 нм с шарнирной областью. в центре. Благодаря гибкости шарнирной области MukB принимает характерную V-образную форму семейства SMC. MukF также имеет тенденцию существовать в виде димера из-за сильного сродства к димеризации между мономерами. С-концевой домен MukF может взаимодействовать с головным доменом MukB, тогда как его центральный домен может взаимодействовать с MukE. Две молекулы MukE и одна молекула MukF связываются друг с другом независимо от MukB с образованием тримерного комплекса (MukE 2 F). Поскольку MukF имеет тенденцию существовать в димерной форме, димеризация MukF приводит к удлиненному гексамерному комплексу (MukE 2 F) 2. В отсутствие АТФ комплекс (MukE 2 F) 2 связывается с головными доменами MukB через С-концевой домен MukF с образованием симметричного комплекса MukBEF (показан слева). Стехиометрия симметричного комплекса B 2 (E 2 F) 2. Связывание АТФ между головными доменами MukB вызывает отщепление одной молекулы MukF и двух молекул MukE. В результате образуется асимметричный комплекс MukBEF стехиометрии B 2 (E 2 F) 1. Поскольку MukF легко димеризуется, димеризация MukF может потенциально соединять две асимметричные молекулы, связанные с АТФ, что приводит к образованию димера димеров со стехиометрией B 4 (E 2 F) 2 (показано справа). Стехиометрия комплекса MukBEF in vivo оценивается как B 4 (E 2 F) 2, что позволяет предположить, что димер димеров является функциональной единицей in vivo. C. Модель экструзии петель димером димеров MukBEF. Димер димера загружается на ДНК (изображена серой линией) через ДНК-связывающие домены MukB. Было показано, что MukB связывает ДНК через свою шарнирную область и верхнюю область своего головного домена. Транслокация комплекса от места его загрузки затем вытесняет петли ДНК. Петли выдавливаются по-скалолазным образом за счет скоординированного открытия и закрытия кольца MukBEF через разъединение головки MukB, которое происходит из-за скоординированного гидролиза АТФ в двух димерах. Темные и голубые круги обозначают события связывания и гидролиза АТФ соответственно. MukE не показан в комплексе для простоты. MukB принадлежит к семейству АТФаз, называемых структурным поддержанием хромосомных белков (SMC), которые участвуют в организации хромосом более высокого порядка у эукариот. Два мономера MukB связываются посредством непрерывного антипараллельного взаимодействия спиральной спирали, образуя жесткий стержень длиной 100 нм. В середине стержня находится гибкая шарнирная область. Благодаря гибкости шарнирной области MukB принимает характерную V-образную форму семейства SMC. Субъединицы, не относящиеся к SMC, ассоциированные с MukB, - это MukE и MukF. Ассоциация закрывает V-образное образование, в результате чего образуются большие кольцеобразные структуры. MukE и MukF кодируются вместе с MukB в одном опероне E. coli. Удаление любой субъединицы приводит к одному и тому же фенотипу, что позволяет предположить, что комплекс MukBEF является функциональной единицей in vivo. ДНК-связывающая активность комплекса находится в субъединице MukB, тогда как MukE и MukF модулируют активность MukB.
Комплекс MukBEF вместе с Topo IV необходим для декатенации и репозиции вновь реплицированных oriC. Роль MukBEF не ограничивается во время репликации ДНК. Он организует и конденсирует ДНК даже в нереплицирующихся клетках. Недавняя конформационная карта хромосом с высоким разрешением штамма E. coli с истощенным MukB показывает, что MukB участвует в формировании взаимодействий ДНК-ДНК на всей хромосоме, за исключением Ter домена. Как запретить MukB действовать в домене Ter? MatP физически взаимодействует с MukB, тем самым предотвращая локализацию MukB в домене Ter. Это проявляется в связывании ДНК MatP и MukB в домене Ter. Связывание ДНК MatP обогащено доменом Ter, тогда как связывание ДНК MukB снижено по сравнению с остальной частью генома. Более того, у штамма, уже лишенного MatP, отсутствие MukB вызывает уменьшение контактов ДНК по всей хромосоме, включая Ter домен. Этот результат согласуется с мнением о том, что MatP вытесняет MukB из Ter домена.
Как комплекс MukBEF функционирует для организации хромосомы E. coli ? Согласно современным представлениям, комплексы SMC организуют хромосомы путем выдавливания петель ДНК. Комплексы SMC перемещаются вдоль ДНК для выдавливания петель цис-способом (на одной и той же молекуле ДНК), при этом размер петель зависит от процессивности комплекса. Комплексы SMC от разных организмов различаются по механизму экструзии петель. Флуоресцентная микроскопия одиночных молекул MukBEF в E. coli предполагает, что минимальная функциональная единица in vivo представляет собой димер димеров. Эта единица образована соединением двух АТФ-связанных комплексов MukBEF посредством MukF-опосредованной димеризации. MukBEF локализуется в клетке в виде 1-3 кластеров, вытянутых параллельно длинной оси клетки. Каждый кластер содержит в среднем ~ 8-10 димеров димеров. Согласно существующей модели, MukBEF вытесняет петли ДНК «скалолазным» способом. Димер димеров высвобождает один сегмент ДНК и захватывает новый сегмент ДНК, не отделяясь от хромосомы. Помимо образования петель ДНК, связь между отрицательной суперспирализацией и функцией MukBEF in vivo вместе со способностью субъединицы MukB сдерживать отрицательные суперспирали in vitro предполагает, что MukBEF организует ДНК, генерируя суперспирали.
Помимо вклада в уплотнение хромосом за счет изгиба, образования мостиков и зацикливания ДНК в меньшем масштабе (~ 1 т.п.н.), NAP участвуют в конденсации и организации ДНК, способствуя протяженным контактам ДНК-ДНК. Два NAP, Fis и HU, стали ключевыми игроками в продвижении дальних контактов ДНК-ДНК, которые происходят по всей хромосоме. Остается изучить, как ДНК-организационная активность Fis и HU, которая хорошо изучена в меньшем масштабе (~ 1 kb), приводит к образованию дальнодействующих взаимодействий ДНК-ДНК. Тем не менее, некоторые из HU-опосредованных взаимодействий ДНК требуют присутствия naRNA4. naRNA4 также участвует в установлении дальних контактов ДНК. HU катализирует некоторые контакты, а не все, что позволяет предположить, что РНК участвует с другими NAP в формировании контактов ДНК. HU также, по-видимому, действует вместе с MukB, способствуя дальнодействующим взаимодействиям ДНК-ДНК. Эта точка зрения основана на наблюдениях, что отсутствие HU или MukB вызывало уменьшение одних и тех же контактов ДНК-ДНК. Неясно, как MukB и HU потенциально действуют вместе, способствуя взаимодействиям ДНК-ДНК. Возможно, эти два белка физически взаимодействуют. В качестве альтернативы, в то время как MukBEF экструдирует большие петли ДНК, HU конденсирует и организует эти петли.
Есть сообщения о том, что функционально связанные гены E. coli физически находятся вместе в трехмерном пространстве внутри хромосомы, даже если они далеки друг от друга генетическим расстоянием. Пространственная близость функционально связанных генов не только делает биологические функции более компартментализированными и эффективными, но также вносит вклад в сворачивание и пространственную организацию нуклеоида. В недавнем исследовании с использованием флуоресцентных маркеров для обнаружения конкретных локусов ДНК изучались попарные физические расстояния между семью оперонами рРНК, которые генетически отделены друг от друга (на целых два миллиона п.н.). Он сообщил, что все опероны, кроме rrn C, находились в физической близости. Удивительно, что исследования 3C-seq не выявили физической кластеризации оперонов rrn, что противоречит результатам исследования, основанного на флуоресценции. Следовательно, необходимы дальнейшие исследования, чтобы разрешить эти противоречивые наблюдения. В другом примере GalR образует сеть взаимодействия сайтов связывания GalR, которые разбросаны по хромосоме. GalR представляет собой регулятор транскрипции регулона галактозы, состоящего из генов, кодирующих ферменты для транспорта и метаболизма сахарной D-галактозы. GalR существует только в одном-двух фокусах в клетках и может самособираться в большие упорядоченные структуры. Следовательно, похоже, что связанный с ДНК GalR мультимеризуется с образованием дальних взаимодействий.
Обычная просвечивающая электронная микроскопия (ПЭМ) химически фиксированных клеток E. coli показала нуклеоид как органеллу неправильной формы. Однако широкопольная флуоресцентная визуализация живых нуклеоидов в 3D выявила дискретную эллипсоидную форму. Наложение фазово-контрастного изображения клетки и флуоресцентного изображения нуклеоида показало близкое сопоставление только в радиальном измерении по всей длине нуклеоида с периферией клетки. Это указывает на радиальное ограничение нуклеоида. Детальное исследование трехмерного флуоресцентного изображения после поперечного сечения, перпендикулярного его длинной оси, дополнительно выявило две глобальные особенности нуклеоида: кривизну и продольные области с высокой плотностью. Исследование хиральности центральной линии нуклеоида путем соединения центра интенсивности каждого поперечного сечения показало, что общая форма нуклеоида искривлена. Распределение интенсивности флуоресценции в поперечных сечениях выявило субструктуру плотности, состоящую из изогнутых областей или пучков с высокой плотностью в центральном ядре и областей с низкой плотностью на периферии. Одним из следствий радиального ограничения является то, что он определяет изогнутую форму нуклеоида. Согласно одной модели, нуклеоид вынужден изгибаться, потому что он заключен в цилиндрическую клетку E. coli, радиус которой меньше ее изгибаемой длины (персистентной длины). Эта модель была подтверждена наблюдениями, что удаление клеточной стенки или ингибирование синтеза клеточной стенки увеличивало радиус клетки и приводило к одновременному увеличению радиуса спирали и уменьшению шага спирали в нуклеоиде.
 Нуклеоид в виде спирального эллипсоида с продольными участками ДНК с высокой плотностью A. Рисунок клетки E. coli с изогнутым нуклеоидом (темно-серый). Изогнутый путь центроидов, обозначенный красными и зелеными точками, подчеркивает изогнутую форму нуклеоида B. Поперечное сечение нуклеоида E. coli, визуализированное HU-mCherry. Интенсивность флуоресценции берется за показатель плотности ДНК и отображается от синего к красному в порядке возрастания.
Нуклеоид в виде спирального эллипсоида с продольными участками ДНК с высокой плотностью A. Рисунок клетки E. coli с изогнутым нуклеоидом (темно-серый). Изогнутый путь центроидов, обозначенный красными и зелеными точками, подчеркивает изогнутую форму нуклеоида B. Поперечное сечение нуклеоида E. coli, визуализированное HU-mCherry. Интенсивность флуоресценции берется за показатель плотности ДНК и отображается от синего к красному в порядке возрастания. Сила расширения, обусловленная связями ДНК-мембрана, по-видимому, действует в противовес силам конденсации, чтобы поддерживать оптимальный уровень конденсации нуклеоида. Исследования клеточного фракционирования и электронная микроскопия впервые показали возможность ДНК-мембранных связей. В настоящее время известно несколько примеров ДНК-мембранных связей. Транссерция - это механизм одновременной транскрипции, трансляции и встраивания растущих мембранных белков, который формирует временные контакты ДНК-мембраны. Было показано, что трансляция двух мембранных белков LacY и TetA вызывает репозиционирование хромосомных локусов по направлению к мембране. Другой механизм нуклеоидно-мембранных связей - это прямой контакт между закрепленными за мембраной регуляторами транскрипции и их сайтами-мишенями в хромосоме. Одним из примеров такого регулятора транскрипции в E. coli является CadC. CadC содержит периплазматический сенсорный домен и цитоплазматический ДНК-связывающий домен. Ощущение кислой среды его периплазматическим сенсорным доменом стимулирует ДНК-связывающую активность CadC, которая затем активирует транскрипцию своих генов-мишеней. Мембранная локализация генов, регулируемых закрепленным за мембраной регулятором транскрипции, еще предстоит продемонстрировать. Тем не менее, ожидается, что активация генов-мишеней в хромосоме этими регуляторами приведет к контакту нуклеоид-мембрана, хотя это будет динамический контакт. Помимо этих примеров, хромосома также специфически прикреплена к клеточной мембране посредством белок-белкового взаимодействия между ДНК-связанными белками, например SlmA и MatP, и дивисомой. Поскольку гены, кодирующие мембранные белки, распределены по всему геному, динамические контакты ДНК с мембраной посредством трансляции могут действовать как сила расширения нуклеоидов. Эта сила расширения будет действовать против сил конденсации для поддержания оптимального уровня конденсации. Образование высококонденсированных нуклеоидов при воздействии на клетки E. coli хлорамфеникола, который блокирует трансляцию, обеспечивает поддержку силы расширения временных контактов ДНК-мембраны, образующихся в результате трансляции. Круглая форма чрезмерно конденсированных нуклеоидов после лечения хлорамфениколом также предполагает роль опосредованных транссерцией контактов ДНК-мембрана в определении эллипсоидной формы нуклеоида.
Нуклеоид может быть четко визуализирован на электронной микрофотографии при очень большом увеличении, где, хотя его внешний вид может отличаться, он отчетливо виден на фоне цитозоля. Иногда видны даже нити того, что считается ДНК. По окрашиванию с красителем Фельген, которые специфически окрашивают ДНК, нуклеоид также можно увидеть под световым микроскопом. ДНК-интеркалирующие пятна DAPI и этидий бромид широко используется для флуоресцентной микроскопии из нуклеоидов. Он имеет неправильную форму и находится в прокариотических клетках.
Изменения в структуре нуклеоида бактерий и архей наблюдаются после воздействия на ДНК повреждающих условий. Нуклеоиды бактерий Bacillus subtilis и Escherichia coli становятся значительно более компактными после УФ-облучения. Формирование компактной структуры в E. coli требует активации RecA посредством специфических взаимодействий RecA-ДНК. Белок RecA играет ключевую роль в гомологичной рекомбинационной репарации повреждений ДНК.
Подобно B. subtilis и E. coli, описанным выше, воздействие на архей Haloferax volcanii стрессов, повреждающих ДНК, вызывает уплотнение и реорганизацию нуклеоида. Уплотнение зависит от белкового комплекса Mre11-Rad50, который катализирует раннюю стадию гомологичной рекомбинационной репарации двухцепочечных разрывов в ДНК. Было высказано предположение, что уплотнение нуклеоидов является частью реакции на повреждение ДНК, которая ускоряет восстановление клеток, помогая белкам репарации ДНК определять местонахождение мишеней и облегчая поиск интактных последовательностей ДНК во время гомологичной рекомбинации.
![]() Эта статья была адаптирована из следующего источника под лицензией CC BY 4.0 ( 2019 ) ( отчеты рецензента ): Subhash Verma; Чжун Цянь; Санкар Л Адхья (декабрь 2019 г.). «Архитектура нуклеоида Escherichia coli». PLOS Genetics. 15 (12): e1008456. DOI : 10.1371 / JOURNAL.PGEN.1008456. ISSN 1553-7390. PMC 6907758. PMID 31830036. Викиданные Q84825966.
Эта статья была адаптирована из следующего источника под лицензией CC BY 4.0 ( 2019 ) ( отчеты рецензента ): Subhash Verma; Чжун Цянь; Санкар Л Адхья (декабрь 2019 г.). «Архитектура нуклеоида Escherichia coli». PLOS Genetics. 15 (12): e1008456. DOI : 10.1371 / JOURNAL.PGEN.1008456. ISSN 1553-7390. PMC 6907758. PMID 31830036. Викиданные Q84825966.