ДНК-кодированная химическая библиотека (DEL ) - это технология для синтез и скрининг в беспрецедентном масштабе коллекций низкомолекулярных соединений. DEL используется в медицинской химии для объединения комбинаторной химии и молекулярной биологии. Целью технологии DEL является ускорением открытия лекарств и, в частности, операций по раннему этапу, как проверка цели и идентификация попаданий.
Технология DEL включает конъюгацию химических соединений или строительных блоков с короткими фрагментами ДНК, которые направляют и контролируют химический синтез. Этот метод позволяет массовое создание и опрос посредством аффинного отбора, обычно на иммобилизованной белковой мишени. Недавно был разработан гомогенный метод скрининга ДНК-кодированных библиотек, в котором используется технология эмульсии вода-в-масле для выделения, подсчета и идентификации индивидуальных комплексов лиганд-мишень в одной пробирке. В отличие от обычных процедур скрининга, как высокопроизводительный скрининг, биохимические анализы не требуются для идентификации связующего, что в принципе позволяет связывающие вещества для широкого диапазона белков, с помощью технологий скрининга. Таким образом, в дополнение к общему открытию молекулярных соединений, специфичных для мишеней, доступность связывающих веществ с фармакологически важными, но пока «не подда обработки» белками-мишенями открывает новые возможности для разработки новых лекарств от болезней, которые пока не поддаются лечению. Отказ от требований от требований оценивать активность совпадений, есть надежда и ожидание, что многие из идентифицированных высокоаффинных продуктов показаны как активные в проверенном анализе выбранных совпадений..
До недавнего времени применения технологии молекулярной эволюции в лаборатории ограничивалось технологиями отображения, включающими В биологические молекулы, в которых малые молекулы приводят к открытию. считалось, что они выходят за рамки этого биологического подхода. Чтобы включить неприродные соединения, такие как малые молекулы, DEL, используйте расширенное использование молекулярной эволюции и естественной эволюции соединений с желаемой активностью функции. ДНК-кодированные химические библиотеки имеют сходство с технологиями биологического дисплея, такими как технология фагового дисплея антител, дрожжевой дисплей, дисплей мРНК и аптамер SELEX. При фаговом дисплее части дисплейелелела, физически связанные с фаговыми, несущими генами, кодирующий прикрепленное антитело, эквивалентно физическому связыванию «фенотипа » (белка) и «генотип » (ген, кодирующий белок). Антитела, отображаемые на фаге, содержат большие библиотек антител, имитировать молекулярную эволюцию: с помощью циклов отбора (на иммобилизованном белке-мишени), амплификации и трансляции. В DEL связывание молекулы с помощью кода ДНК идентифицировать связывающие молекулы. Библиотеки DELETE подвергают тестовой процедуре аффинной селекции на выбранном иммобилизованном целевом белке, после чего несвязывающие вещества могут быть амплифицированы с помощью их ДНК (например, секвенирования). В эволюционных технологиях DEL (см. Ниже) совпадения могут быть дополнительно обогащены путем проведения раундов отбора, ПЦР-амплификации и трансляции по аналогии с системами биологического дисплея, такими как фаговый дисплей антител. Это дает возможность работать с большой библиотекой.
«Синтезировать многокомпонентную смесь соединений в одном процессе и просеивать их также в одном процессе». Это принцип комбинаторной химии, изобретенный профессором Фуркой А. (Университет Этвёша Лоранда, Будапешт, Венгрия) в 1982 г. и описал его, включая метод набора комбинаторных библиотек и метод деконволюции, в документе, нотариально заверенном в том же году. Мотивы, которые привели к изобретению, были опубликованы в 2002 году. DEL - это комбинаторные библиотеки, кодируемые ДНК (DECL), и комбинаторный принцип явно преобладает в их применении.
 Фиг. 1 ДНК-кодированная библиотека, отображающая химические соединения Схематическое изображение ДНК-кодируемой библиотеки, отображающую химические соединения, присоединенные к олигонуклеотидам. а) Библиотека, созданная путем «поэтапной комбинаторной» сборки, представляющая единственный олигонуклеотид, ковалентно связанный с предполагаемой связывающей молекулами. б) Конструирование библиотеки в режиме «комбинаторной самосборки» (E ncoded S elf- A ssembly C химическая библиотека). Множественные сплетенные олигонуклеотиды демонстрируют ковалентно связанные связывающие молекулу
Фиг. 1 ДНК-кодированная библиотека, отображающая химические соединения Схематическое изображение ДНК-кодируемой библиотеки, отображающую химические соединения, присоединенные к олигонуклеотидам. а) Библиотека, созданная путем «поэтапной комбинаторной» сборки, представляющая единственный олигонуклеотид, ковалентно связанный с предполагаемой связывающей молекулами. б) Конструирование библиотеки в режиме «комбинаторной самосборки» (E ncoded S elf- A ssembly C химическая библиотека). Множественные сплетенные олигонуклеотиды демонстрируют ковалентно связанные связывающие молекулу Концепция кодирования ДНК была впервые описана в теоретической статье Бреннера и Лернера в 1992 году, в результате чего было предложено связать каждую молекулу химически синтезированного объекта с конкретным олигонуклеотидная последовательность, сконструированная управляемая последовательность, и использование этой кодирующей метки для идентификации и обогащения соединений. В 1993 году практическая реализация этого была представлена первая С. Бреннером и К. Янда, а также группа М.А.Гэллопа. Бреннер и Джанда предложили создать отдельные кодируемые члены библиотеки посредством чередующегося параллельного комбинаторного синтеза гетерополимерного химического соединения и реализации олигонуклеотидной системы на одной и той же грануле на основе разделения и (см.).
незащищенная ДНК ограничена узким интервалом обычных условий реакции до конца 1990-х годов обеспечивает ряд альтернативных стратегий кодирования (т. Е. мечение соединения на основе MS, код пептида, галогенароматическое мечение, кодирование вторичными аминами, полупроводниковыми устройствами.), В основном, чтобы избежать неудобного твердофазного синтеза ДНК и создания легко просматриваемых комбинаторных библиотек с высокой пропускной способностью способностью. Однако избирательная амплифицируемость ДНК облегчает скрининг библиотек и незаменимую кодирование библиотек такого беспрецедентного размера. Следовательно, в начале 2000-х годов ДНК-комбинаторная химия пережила возрождение.
В начале тысячелетия появилось несколько независимых разработчиков технологии DEL. Эти технологии можно разделить на две основные категории: технологии DEL, не основанные на эволюции, и технологии DEL, способные к молекулярной эволюции. Первая категория выигрывает от возможности готовые реагенты и, следовательно, позволяет довольно просто создать библиотеку. Попадания можно идентифицировать с помощью секвенирования ДНК, однако трансляция ДНК и, следовательно, молекулярная методика невозможны. Подходы с разделением и объединением, разработанные исследователями из Praecis Pharmaceuticals (в настоящее время принадлежащей GlaxoSmithKline), Nuevolution (Копенгаген, Дания) и технологии ESAC, разработанной в лаборатории профессора Д. Нери (Институт фармацевтических наук, Цюрих, Швейцария) подпадают под эту категорию.. Технология ESAC отличается тем, что является комбинаторным подходом к самосборке, который напоминает попаданий на основе фрагментов (рис. 1b). Здесь отжиг ДНК позволяет отбирать дискретных комбинаций строительных блоков, но между ними не происходит реакции. Примерами технологий DEL, основанных на эволюции, ДНК-маршрутизация, разработанная профессором Д. Халпин и проф. Харбери (Стэнфордский университет, Стэнфорд, Калифорния), синтез на основе ДНК, шаблон профессора Д. Лю (Гарвардский университет, Кембридж, Массачусетс) и коммерциализируется Ensemble Therapeutics (Кембридж, Массачусетс) и YoctoReactor Technology. разработан и коммерциализирован компанией Vipergen (Копенгаген, Дания). Эти технологии более подробно продвижение ниже. Синтез на основе ДНК и технологий YoctoReactor требует предварительной конъюгации химических блоков (BB) с ДНК-олигонуклеотидным тегом перед сборкой библиотеки, поэтому перед сборкой библиотеки требуется дополнительная предварительная работа. Кроме того, ДНК-метки могут генерировать генетический код для синтезированных соединений, и возможна искусственная трансляция генетического кода: то есть BB отозваны с помощью генетического кода, усиленный ПЦР, и соединения библиотеки могут быть регенерированы. Это, в свою очередь, позволяет применять принцип дарвиновского естественного отбора и эволюции к отбору малых молекул по аналогии с биологическими системами отображения; через этапы отбора, расширения и перевода.
Комбинаторные библиотеки - это особые многокомпонентные смеси, которые синтезируются в одностадийном процессе. Они отличаются от набора отдельных соединений, а также от соединений, полученных параллельным синтезом. Комбинаторные библиотеки имеют важные особенности.
″ В их синтезе используются смеси. Использование смесей очень высокой эффективности процесса. Оба реагента могут быть смесями, но по практическим соображениям используется процедура раздельного смешивания: одна смесь разделяется на части, которые соединяются с BB. Смеси настолько важны, что не существует комбинаторной библиотеки без использования смеси в синтезе, и если смесь используется в процессе, неизбежно образуется комбинаторная библиотека.
″ Компоненты библиотек должны присутствовать примерно в равных молярных количествах. Чтобы добиться этого как можно точнее, смеси делятся на равные части, и после объединения необходимо тщательное перемешивание.
″ Так как состав компонентов неизвестен, при скрининге необходимо использовать методы деконволюции. По этой причине были разработаны методы кодирования. Кодирующие молекулы прикреплены к шарикам твердой подложки, которые связаны между собой BB и их последовательность. Один из этих методов - кодирование олигомерами ДНК.
″ Замечательной особенностью комбинаторных библиотек является то, что вся смесь соединений может быть проверена в одном процессе.
Синтезатор и скрининг являются очень эффективными процедурами, использование библиотек в фармацевтических исследованиях приводит к огромной экономии.
При твердофазном комбинаторном синтезе в каждой грануле образует только одно соединение. По этой причине компонентов в библиотеке может быть количество валиков твердой опоры. Это означает, что количество компонентов в такой библиотеке ограничено. Это ограничение было полностью снято Харбери и Халпином. При их синтезе DEL твердый носитель не используется, а BB присоединяется непосредственно к кодирующим олигомерам ДНК. Этот новый подход неограниченно увеличивать количество компонентов комбинаторных библиотек, кодируемых ДНК (DECL).
Чтобы применить комбинаторную химию для синтеза кодируемых ДНК химических библиотек, был применен подход с разделением пула.. Первоначально набор уникальных ДНК- , каждый из которых содержит конкретную кодирующую последовательность, химически конъюгирован с набором конкретных молекул. Следовательно, соединения олигонуклеотид -конъюгата смешивают («Пул») и разделяют («Разделить») на ряд групп (m ). В подходящих условиях второй набор строительных блоков (м) связывают с первым, дополнительным олигонуклеотид, который кодирует вторую модификацию, вводит ферментативно перед повторным смешиванием. Эти этапы разделения «- и - пула» могут повторяться несколько раз (r ), увеличивая на каждом этапе размер библиотеки комбинаторным способом (то есть (nx m)). Были использованы кодирования библиотек, пептидные нуклеиновые кислоты, данные разделения и объединения. Преимущество PNA-кодирования состоит в том, что химический процесс можно проводить с помощью стандартного SPPS.
 Рис. 3 ДНК-кодируемая библиотека с помощью «Split - - Pool ступенчатого связывания кодирующих фрагментов ДНК с органическими молекулами. Начальный набор многофункциональных строительных блоков (FGn представляет различные ортогональные функциональные группы) ковалентно конъюгирован к соответствующему кодирующему олигонуклеотиду и прореагировала по принципу разделения и пула на функциональную группу (FG1 в красном цвете) с подходящим набором реагентов. После ферментативного кодирования инициируется следующий раунд разделения пула. стадия вторая функциональная группа (FG2 в синем) подвергается дополнительной стадии реакции с другим набором подходящих реагентов. Идентичность окончательной модификации может быть еще раз подтверждена ферментативным кодированием ДНК с помощью дополнительного олигонуклеотида, несущего конкретную кодирующую область.
Рис. 3 ДНК-кодируемая библиотека с помощью «Split - - Pool ступенчатого связывания кодирующих фрагментов ДНК с органическими молекулами. Начальный набор многофункциональных строительных блоков (FGn представляет различные ортогональные функциональные группы) ковалентно конъюгирован к соответствующему кодирующему олигонуклеотиду и прореагировала по принципу разделения и пула на функциональную группу (FG1 в красном цвете) с подходящим набором реагентов. После ферментативного кодирования инициируется следующий раунд разделения пула. стадия вторая функциональная группа (FG2 в синем) подвергается дополнительной стадии реакции с другим набором подходящих реагентов. Идентичность окончательной модификации может быть еще раз подтверждена ферментативным кодированием ДНК с помощью дополнительного олигонуклеотида, несущего конкретную кодирующую область. Многообещающая стратегия создания ДНК-кодированных многофункциональных строительных блоков , ковалентно конъюг с олигонуклеотидом, служащим как «основная структура» для алгоритма библиотеки. По принципу «объединить и разделить» набор многофункциональных карсов претерпевает ортогональные реакции с рядом подходящих реактивных партнеров. После стадии реакции модификация кодируется ферментативным добавлением каждой ДНК к исходной «ядерной структуре» ДНК. Использование N-защищенных аминокислот, ковалентно связанного фрагмента ДНК, позволяет после подходящей стадии снятия защиты образование дополнительной амидной с рядом карбоновых кислот или восстановительным аминированием. с альдегидами. Аналогичным образом, диен карбоновые кислоты, используемые в каркасов для конструирования библиотеки на 5'-конце амино-модифицированного олигонуклеотида, могут быть подвергнуты реакции Дильса-Альдера с различными производные малеимида. После завершения желательной стадии реакции идентичность химического фрагмента, добавленного к олигонуклеотиду, устанавливается посредством отжига частично комплементарного олигонуклеотида и последующего Кленова вставка ДНК-полимеризация с получением двухцепочечного фрагмента ДНК. Стратегии синтеза и кодирования, описанные выше, позволяют легко создать библиотеки, кодируемые ДНК, размером до 10 соединений-членов, несущих два набора «строительных блоков». Однако может быть предусмотрено поэтапное добавление по крайней мере трех независимых программных групп к функциональному блоку ядра для конструирования и кодирования большой ДНК-кодируемой библиотеки (включающей до 10 соединений). (Рис.2 )
 Рис. 4 Обзор технологии библиотеки ESAC Небольшие органические молекулы связаны с 5'-амино Библиотека ESAC может сообщить в формате единственного фарфора (а), при созревании аффинности считывающих сообщающих веществ (b) или ново отборных молекул самосборки подбиблиотек в формате двухцепочечной системы Библиотека ESAC в выбранном формате используется в процедуре отбора и принятия (e). белка выбора (ii) и отмывки несвязанных молекул (iii), олигонуклеотидные коды связывающих соединений амплифицируются с помощью ПЦР и сравниваются с библиотекой без отбора на олигонуклеотидные микромассива (iv, v). по необходимости) с подходящими каркасами (vi).
Рис. 4 Обзор технологии библиотеки ESAC Небольшие органические молекулы связаны с 5'-амино Библиотека ESAC может сообщить в формате единственного фарфора (а), при созревании аффинности считывающих сообщающих веществ (b) или ново отборных молекул самосборки подбиблиотек в формате двухцепочечной системы Библиотека ESAC в выбранном формате используется в процедуре отбора и принятия (e). белка выбора (ii) и отмывки несвязанных молекул (iii), олигонуклеотидные коды связывающих соединений амплифицируются с помощью ПЦР и сравниваются с библиотекой без отбора на олигонуклеотидные микромассива (iv, v). по необходимости) с подходящими каркасами (vi). Encoded S elf- A ssemble C hemical (ESAC ) основы в принципе, что две подбиблиотеки размером x членов (например, 10), содержат константный комплементарный домен гибридизации, может дать библиотеку комбинаторного дуплекса ДНК после гибридизации со сложностью x одинаково представленных элементов библиотеки (например, 10). Каждый член подбиблиотеки может состоять из олигонуклеотида, содержащего вариабельную кодирующую область, фланкированную константную последовательность ДНК, несущую подходящую химическую модификацию на конце олигонуклеотида. Подбиблиотеки ESAC Мои книги по меньшей мере в четырех различных вариантах осуществления.
Предпочтительные связывающие вещества, выделенные в результате селекции на основе аффинности, могут быть амплифицированы с помощью ПЦР и декодируются на комплементарных олигонуклеотидных микрочипах или путем конкатенации кодов, субклонирования и секвенирования. В конечном итоге строительные блоки можно конъюгировать с использованием подходящих линкеров, чтобы получить лекарственное соединение с высоким сродством. Характеристики линкера (например, длина, гибкость, геометрия, химическая и растворимость) влияние на сродство природы связывания и химические свойства полученного связующего. (Фиг.3 )
Биопэннинг эксперименты с HSA из 600-членной библиотеки ESAC позволили выделить 4- (п-иодфенил) бутановую группу. Портативные связывающие альбумин молекулы и альбуфлуора, недавно разработанное флуоресцеина ангиографического контрастного вещества, которое было в настоящее время проходит клиническую оценку.
Технология ESAC для выделения сильнодействующих ингибиторов бычьего трипсина и для идентификации новых ингибиторов (MMP-3 ), матрица металлопротеиназа участвующая в физиологических, так и в патологических процессов, ремоделирования тканей, а также патологических процессов, таких как артрит и метастаз.
В В 2004 году Д. Халпин и П. Харбери представил новый интригующий метод создания библиотек, кодируемых ДНК-конъюгированных шаблонов для кодирования, так и для программирования и нфраструкту. ры синтез компонентов библиотеки по принципу «split - - pool». Дизайн Халпина и Харбериил чередовать раунды отбора, ПЦР-амплификации и диверсификации с использованием легких молекул, в полном аналогии с технологией фагового дисплея. Аппарат ДНК-маршрутизации состоит из нескольких соединенных столбцов, несущих связанные со смолой антикодоны, которые могут секвенциально разделять популяцию ДНК-матриц на пространственно местоположениях посредством гибридизации. В соответствии с этим протоколом разделения и объединения была создана пептид комбинаторная библиотека ДНК, кодируемая из 10 членов.
 Фиг. 2 ДНК-кодируемая библиотека с помощью ДНК-шаблонного синтеза Библиотека олигонуклеотидов (т.е. 64 различных олигонуклеотидов), содержащая три кодирующие области, была гибридизирована с библиотекой конъюгатов соединение-реагент-олигонуклеотид (т. е. 4 реагента олигонуклеотидные конъюгаты), способные к спариванию с начальным кодирующим доменом матричного олигонуклеотида. После переноса соединения на соответствующий олигонуклеотидную матрицу цикл повторяли желаемое количество с дополнительным набором конъюгатов соединение-носитель-олигонуклеотид (т.е. два цикла четырьмя конъюгатами соединение-носитель-олигонуклеотид на цикл). Впоследствии был выполнен функциональный отбор и последовательность связывающей матрицы амплифицировали с помощью ПЦР. Таким образом, ДНК-секвенирование идентифицировать связывающую молекулу.
Фиг. 2 ДНК-кодируемая библиотека с помощью ДНК-шаблонного синтеза Библиотека олигонуклеотидов (т.е. 64 различных олигонуклеотидов), содержащая три кодирующие области, была гибридизирована с библиотекой конъюгатов соединение-реагент-олигонуклеотид (т. е. 4 реагента олигонуклеотидные конъюгаты), способные к спариванию с начальным кодирующим доменом матричного олигонуклеотида. После переноса соединения на соответствующий олигонуклеотидную матрицу цикл повторяли желаемое количество с дополнительным набором конъюгатов соединение-носитель-олигонуклеотид (т.е. два цикла четырьмя конъюгатами соединение-носитель-олигонуклеотид на цикл). Впоследствии был выполнен функциональный отбор и последовательность связывающей матрицы амплифицировали с помощью ПЦР. Таким образом, ДНК-секвенирование идентифицировать связывающую молекулу. В 2001 году Дэвид Лю и его сотрудники показали, что комплементарные ДНК олигонуклеотиды выключены для облегчения синтетических факторов, которые неэффективны в растворе при низкой <содержит14>. ДНК-гетеродуплекс использовали для ускорения реакции между химическими фрагментами, отображаемыми на концах двух цепей ДНК. Кроме того, было показано, что «эффект близости» ускоряет бимолекулярную реакцию того, не зависит от расстояния (по крайней мере, на 30 нуклеотидов ). В соответствии с запрограммированной последовательностью олигонуклеотиды, несущие одну группу химического реагента, были гибридизированы с комплементарными производными олигонуклеотидов, несущими другую реактивную химическую группу. Близость, обеспечиваемая гибридизацией ДНК, увеличивает эффективную молярность реакционных реагентов, присоединенных к олигонуклеотидам, позволяя протекать желаемую реакцию даже в водной среде при навыках, которые на несколько порядков ниже необходимых. для обычной органической реакции без ДНК-шаблона. Используя ДНК-шаблон и запрограммированный синтез, Лю и его сотрудники создали 64-членную кодированную ДНК-библиотеку, состоящую из макроциклов.
YoctoReactor (yR) - это трехмерный подход, основанный на соединении, который использует самособирающуюся природу олигонуклеотидов ДНК в 3-, 4- или 5-сторонние соединения, чтобы направить синтез малых молекул в центре соединения. Рисунок 5 представляет базовую концепцию четырехстороннего соединения ДНК.
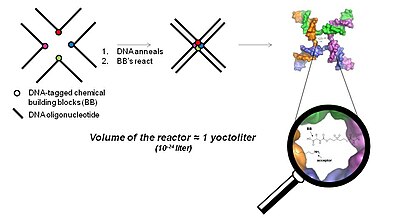 Фиг. 5 Фундаментальный принцип YoctoReactor. Центр 3-, 4- и 5-сторонних соединений ДНК (здесь показан четырехсторонний переход) становится реактором в масштабе йоктолитров, где синтез малых молекул облегчается в том, что было названо YoctoReactor (yR). Цветными кружками изображены химические строительные блоки (BB), прикрепленные к черным тщательно разработанным олигонуклеотидам ДНК (линии). После отжига ДНК BB сближаются с помощью соединения ДНК.
Фиг. 5 Фундаментальный принцип YoctoReactor. Центр 3-, 4- и 5-сторонних соединений ДНК (здесь показан четырехсторонний переход) становится реактором в масштабе йоктолитров, где синтез малых молекул облегчается в том, что было названо YoctoReactor (yR). Цветными кружками изображены химические строительные блоки (BB), прикрепленные к черным тщательно разработанным олигонуклеотидам ДНК (линии). После отжига ДНК BB сближаются с помощью соединения ДНК. Центр соединения ДНК составляет объем порядка йоктолитра, отсюда и название YoctoReactor. Этот объем производит реакцию одной молекулы, дающая реакционная способность в диапазоне высоких мМ. Эффективная система, способствующая действию ДНК, ускоряет химические реакции.
На рисунке 6 показано создание библиотеки yR с использованием 3-стороннего соединения ДНК.
Таким образом, химические строительные блоки (BB) присоединяются через расщепляемые или нерасщепляемые линкеры к три типа биспецифических олигонуклеотидов ДНК (олиго-BB), представляющих каждое плечо года. Чтобы облегчить синтезаторным способом, олиго-ВВ сконструирована таким образом, чтобы ДНК-ДНК была создана, чтобы вызвать самосборку ДНК в 3, код для присоединенного ВВ на дистальном олиго (цветные линии) и (б) области последовательной ДНК (черная линия) - стороннее соединение (независимо от BB) и последующую химическую реакцию. Химические реакции выполняются поэтапно, и после каждого этапа ДНК лигируют, а продукт очищают электрофорезом в полиакриамидном геле. Расщепляемые линкеры (BB-ДНК) используются для всех позиций, кроме одного, что дает библиотеку молекул с одной ковалентной связью с кодом ДНК. В таблице 1 показано, как библиотеки разных размеров могут быть созданы с использованием технологии yR.
 Таблица 1. Размер библиотеки YoctoReactor. Размер библиотеки yR - это функция количества различных функциональных олигонуклеотидов, используемых в каждой позиции и количестве позиций в соединении ДНК.
Таблица 1. Размер библиотеки YoctoReactor. Размер библиотеки yR - это функция количества различных функциональных олигонуклеотидов, используемых в каждой позиции и количестве позиций в соединении ДНК. YR подход к проектированию необходимого участка реакции в отношении как (а) расстояния между реагентами, так и (б) окружения, окружающей среды реакции. Сокращенная связь между кодом и BB на олиго-BB фрагменты, которые комбинаторно смешаны в одном горшке, дает высокую точность кодирования библиотеки. Код синтезированных продуктов, кроме того, не задан заранее, а скорее комбинаторно собирается и синтезируется синхронно с врожденным продуктом.
Недавно был разработан гомогенный метод скрининга йоктореакторных библиотек (yR), использующий технологию эмульсии вода-в-масле для выделения индивидусов лиганд-мишень. Называемое обогащением связующей ловушки (BTE), лиганды к белку-мишени идентифицируются путем улавливания связывающих пар (меченого ДНК белка-мишени и yR-лиганда) в каплях эмульсии во время доминирующей кинетики диссоциации. После захвата ДНК-мишень и лиганд соединяются путем лигирования, таким образом сохраняя информацию о связывании.
В дальнейшем идентификация совпадений - это, по сути, упражнение по подсчету: информация о событиях связывания расшифровывается путем секвенирования и подсчета объединенной ДНК - селективные связывающие элементы подсчитываются с большей степенью, чем случайные связывающие. Это возможно, потому что случайный захват мишени и лиганда «разбавляется» большим количеством капель воды в эмульсии. Низкий уровень шума и фонового сигнала, характерный для BTE, объясняется «разбавлением» случайного сигнала, отсутствием поверхностных артефактов и высокой библиотеки метода скрининга. Скрининг проводится методом одной пробирки. Биологически активные попадания выявляются в одном раунде BTE, характеризующемся низким уровнем ложноположительных результатов.
BTE имитирует неравновесную природу взаимодействия лиганд-мишень in vivo и предлагает уникальную возможность скрининга на предмет специфических лигандов на основе времени пребывания лиганд-мишень, поскольку образует эмульсию, которая захватывает связывающий комплекс во время фазы динамической диссоциации.
После выбора из химических библиотек, кодируемых ДНК, стратегии декодирования для быстрой идентификации специфических связывающих соединений имеет решающее значение для дальнейшего развития Технология DEL . До сих пор секвенирование по Сэнгеру на основе декодирования, микроматрица методология и высокопроизводительное секвенирование представляет основные методологии для декодирования ДНК-кодированной библиотеки. выбор.
Хотя многие авторы неявно предусматривают традиционное декодирование на основе секвенирования по Сэнгеру, количество кодов для секвенирования просто зависит от сложности библиотеки это определенно нереальная задача для традиционного подхода секвенирования по Сэнгеру. Тем не менее, реализация секвенирования по Сэнгеру для декодирования ДНК-кодированных химических библиотек высокопроизводительным способом описана первой. После отбора и ПЦР-амплификации ДНК-тегов соединений библиотеки были созданы конкатамеры, содержащие несколько кодирующих последовательностей, и лигированы в вектор. После секвенирования по Сэнгеру репрезентативного числа образся колоний были выявлены частоты кодов, присутствующих в кодируемой ДНК образце библиотеки до и после отбора.
ДНК микроматрица - это устройство для высокопроизводительных исследований, широко используемое в молекулярной биологии и в медицине. Он состоит из упорядоченной серии микроскопических положений, содержащих несколько пикомолей олигонуклеотидов, несуществующую последовательность ДНК. Это может быть короткий участок гена или другого элемента ДНК, который используется в зондов для гибридизации образца ДНК или РНК в подходящих условиях. Гибридизация зонда с мишенью обычно выявляется и количественно определяется с помощью флуоресцентного обнаружения меченных флуорофором мишеней для определения относительного количества целевой нуклеиновой кислоты. Микроматрица была успешно для успешного декодирования библиотек, кодируемых ESAC ДНК, и библиотек, кодируемых PNA. Кодирующие олигонуклеотиды, представляющие отдельные химические соединения в библиотеке, наносятся пятнами и химически связываются на предметных стеклах микроматрицы с использованием робота BioChip Arrayer. Затем олигонуклеотидные теги связывающих соединений, выделенных в результате отбора, амплифицируют ПЦР с использованием флуоресцентного праймера и гибридизуют на слайд ДНК- микроматрицы. После этого микроматрицы анализируются с использованием лазерного сканирования, и интенсивности пятен обнаруживаются и количественно оцениваются. Обогащение предпочтительными связывающими соединениями выявляется при сравнении интенсивности пятен на слайде ДНК- микроматрицы до и после отбора.
Согласно сложность кодируемой ДНК химической библиотеки (обычно от 10 до 10 членов), обычное декодирование на основе секвенирования Сэнгера вряд ли будет применимо на практике из-за высокой стоимости секвенирования на основание и Это утомительная процедура. В технологиях высокопроизводительного секвенирования использовались стратегии, которые распараллеливают процесс секвенирования, заменяя использование капиллярного электрофореза и создавая тысячи или миллионы последовательностей одновременно. В 2008 году была описана первая реализация метода высокопроизводительного секвенирования, первоначально разработанного для секвенирования генома (то есть «технология 454 ») для быстрого и эффективного декодирования кодированной ДНК химической библиотеки. в составе 4000 соединений. Это исследование привело к идентификации новых химических соединений с субмикромолярными константами диссоциации по отношению к стрептавидину и определенно показало возможность создания, выполнения отбора и декодирования кодированных ДНК библиотек, содержащих миллионы химических соединений.